И вот не прошло и полтора года как я добрался собрать третичную структуру тРНК. Напомню, что ранее писал статью на эту тему на хабре Развитие RNAInSpace… . Надо сказать, что около года я этим не занимался, но за это время вышла вторая моя научная статья на эту тему Применение теории игр для задачи сворачивания рибонуклеиновых кислот (это для тех кто захочет поговорить об этом профессионально). Но вот недавно я можно сказать получил третичную структуру тРНК и сверил её с образцом имеющимся в базе данных (PDB), который получен биологическими методами (кристалография).
Под катом рисунки 3D-структуры тРНК, пояснения и планы на будущие…
Третичная структура тРНК — результаты
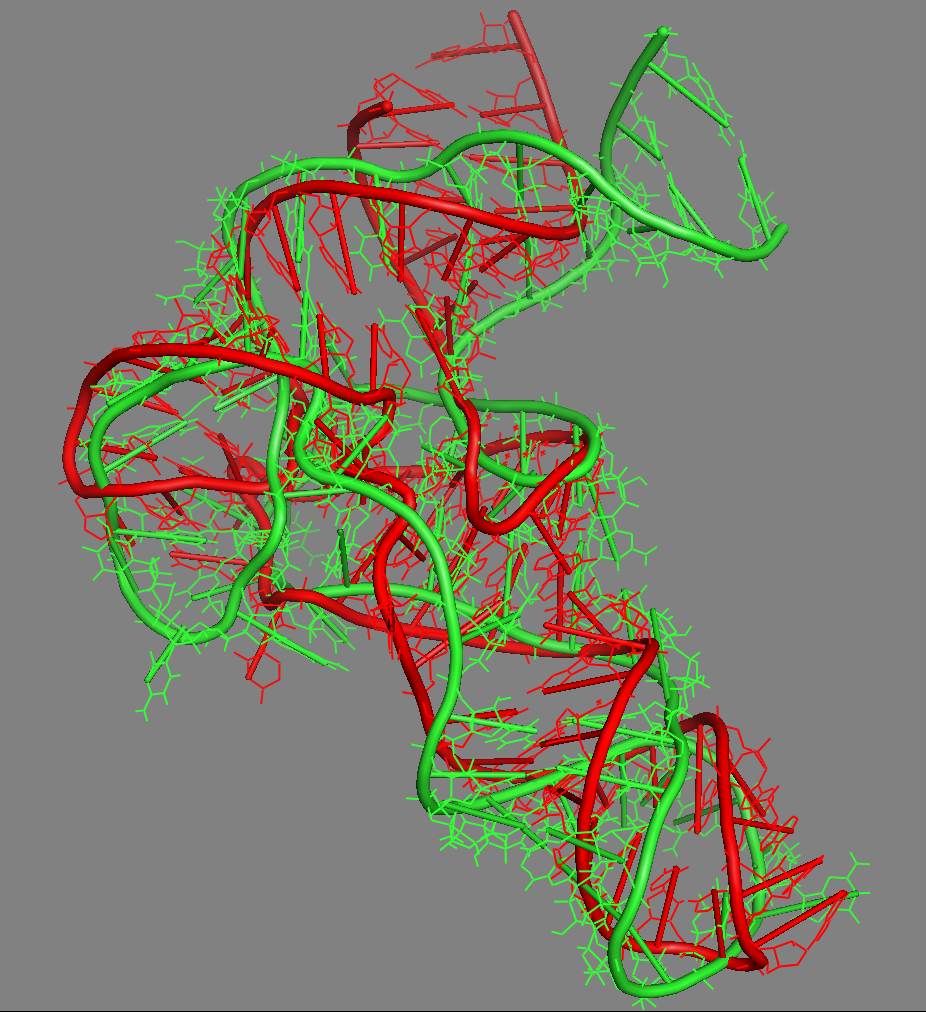
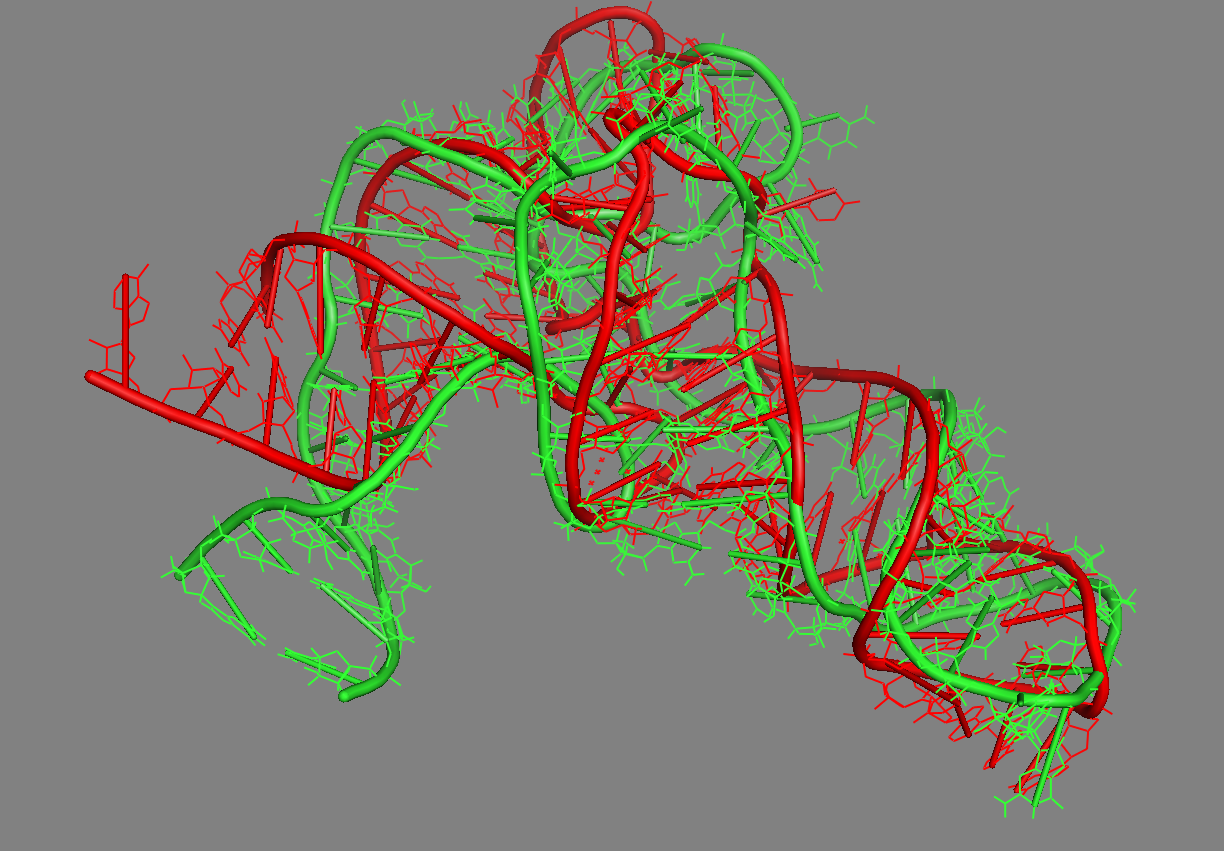
Можно было бы сделать ролик сворачивания, но я поленился — он мало что показывает, как пример начала можно посмотреть этот, потом этот, и потом сворачивание превращает цепь в тРНК, изображенную на рисунке.
На рисунках тРНК с двух углов зрения. Зеленная это модель полученная мной, красная это модель из базы PDB. Теперь я могу сказать специалистам, что RMSD = 6,71 (это мера схожести двух моделей). Как можем видеть общий профиль практически совпадает. Так же в моей модели образованы практически все стандартные водородные связи и близки к образованию неканонические водородные связи.
Надо отметить (напомнить, если кто-то уже читал мои статьи), что третичная модель получается у меня только на основании первичной структуры (т.н. de novo), при возможности спрогнозировать места водородных связей и найти критически важные места стэкинга. Если будет интерес, готов пояснить детали и обсудить эти результаты.
Закрытие сезона
Доведя это направление моей деятельности до некоторого логического заключения — этой статьей я хотел бы закрыть серию статей, которые я написал на Хабре. По сути своих целей я добился. И тут об этом тезисно расскажу:
1. Первая статья в интернете датируется 2009 годом. В ней задача фолдинга ставится в духе кибернетических идей.
2. Далее я пытался развить открытый проект в Викиверситете .
Основной тезис был следующий "можно получить серьезные результаты, зная лишь определенный минимум и не имея профильного образования ни в биологии, ни в физике, ни в химии". Теперь у меня нет сомнений, что серьезные результаты я получил, а метод которым я получил превосходит все прочие методы, существующие на данный момент.
Так что господа не бойтесь начинать, на своем пути вы встретите много противодействия и критики тех кто мало в чем разбирается, но готов показать свою эрудированность. При наличии результатов им придется отступить.
3. Мне пришлось отказаться от многих современных подходов в этом направлении, порой возникало даже ощущение, что методы применяют не для того, чтобы решить задачу, а чтобы показать как работает тот или иной метод. и если вначале я возлагал надежды на какие то методы, в том числе методы искуственного интеллекта, то оказалось что они не годятся. Годится лишь общая идеология теории игр и агентного подхода. А так все сводится к определенным эвристикам в нахождении целевой функции (конечно если говорить подробнее, есть мелкие вкусности в разработанных мной алгоритмах — но это не для этой статьи — не тот уровень погружения в проблематику)
4. Две статьи в реферируемых журналах — лично для меня достаточно на эту тему. Спасибо за внимание :)
5. По сути я разработал метод и подход, теперь дело за техникой и последователями.
6. А далее, я прихожу к вопросу «ради чего и зачем»? Об этом в следующем разделе.
«Отличие живого от не живого»
Еще в той первой статье ответ на вопрос зачем изучать трехмерную структуру РНК был дан (это помимо того, что это интересно само по себе, и может быть полезно биологам)
мы имеем четкую биологическую задачу: «Выяснить какие именно и на сколько изменения в трехмерной структуре 50-100 нуклеотидной цепи РНК принципиально влияют на то, чтобы эта цепь РНК являлась рибозимом». Другими словами, какие мутации рибозима улучшают или ухудшают возможность саморепликации, вплоть до их отсутствия. И популяризуя – это и будет детальный ответ на вопрос, чем отличается живое от не живого
Конечно, оглядываясь теперь это несколько наивно. Но тем не менее несет определенный смысл. Попробую пояснить.
Еще ранее я не раз отмечал, что современная теория выравнивания последовательностей по сути ошибочна, она позволяет по сути подгонять результаты, а не получать истиную картину. Так же я писал, что аннотирование в биологических базах содержит множество ошибок Геномы секвенированных организмов — ошибки в базах, и те кто там работают были вынуждены с этим согласится.
Теперь оглядываясь назад, могу сказать, что тогда я не зная по сути биоинформатики в своей первой статье «сделал ставку» на т.н. Структурное выравнивание. Это такое нахождение генов в геноме, и последующие сравнение геномных последовательностей, которое учитывает НЕ мутации отдельных нуклеотидов и их статистику, а ориентируется на третичную структуру функционально похожих генов.
Действительно, теперь мой подход получения третичной структуры позволяет судить о том, сможет ли определенная нуклеотидная последовательность свернутся в ту или иную структуру. А это означает, что можно понять какие части нуклеотидной последовательности должны быть консервативными, а в каких возможны мутации.
Вся эта информация, которая действительно влияет на возможность функционирования той же тРНК, или рибозима или любой другой структуры РНК — при простом анализе (выравнивании) не используется, а значит там однозначно будут ошибки, которые будут даже не заметны для иследователя, который не обращает внимание на функциональность третичной структуры. А статистический подход, который повсеместно для этого сейчас используется, еще больше затемнит этот вопрос.
И вот, теперь когда мы знаем (приблизительно) третичную структуру — мы можем построить, назовем это — функциональный профиль, например, тРНК. И после этого, и только после этого — сможем найти с достаточной точность в ДНК места расположения всех тРНК.
Но построить этот функциональный профиль не так и просто. Оказывается у нас мало 100% консервативных участков — практически все может меняться в абсолютном значении. Чтобы это понять рассмотрим пример с тРНК.
Вот давайте сравним две тРНК:
gcgcggauagcucagucgguagagcaggggauugaaaauccccguguccuugguucgauuccgaguccgcgc
gcggauuuagcucaguugggagagcgccagacugaagucuggagguccuguguucgauccacagaauucgca
попробуйте выравнить эти две тРНК и сказать чем же они отличаются? В реальности проблема много хуже — эти последовательности не выделены, как в данном примере — они находятся среди милионов подобных же знаков g-c-a-u. И мы не знаем где нужные нам тРНК.
Можно конечно заниматься ерундой и выравнивать эти знаки, делая предположения где разрывы, а где вставки произошедшие при мутировании.
Но можно поступить проще, давайте найдем водородные связи, для начала хотя бы классические. Получим:
(((((((..((((........)))).(((((((...))))))).....(((((.......))))))))))))
(((((((..((((........)))).((((((....)))))).....(((((.......)))))))))))).
Не правда ли становится веселее? Оказывается разница то уже не столь большая. Надо сделать допуски на плюс минус 1-3 точки (неспаренные нуклеотиды) и 1-3 пары скобки (спаренные водородной связью нуклеотиды). Для получения большей точности можно будет найти и соответствие неканоннических водородных связей (которые и стабилизируют структуру на 3D уровне).
Конечно по прежднему сложно находить эти структуры среди милионов знаков g-c-a-u. Но и тут есть ориентир. Разделим задачу на части, и поищем не все тРНК, а те которые приносят Фенилаланин. А раз так мы точно знаем, что в центре находится последовательность gaa. Тогда мы можем искать все такие последовательности в геноме у который в середине gaa, а также имеются соответствующий профиль:
(((((((..((((........)))).(((((((gaa))))))).....(((((.......))))))))))))
(((((((..((((........)))).((((((gaa.)))))).....(((((.......)))))))))))).
с допустимым пределами в структуре.
Вот этим я собираюсь заняться в ближайшее время — достоверно найти все тРНК в секвенированных геномах бактерий. Может кто-то захочет в этом поучаствовать — приглашаю.
Автор: tac






