Сергей Науменко, научный сотрудник лаборатории эволюционной геномики факультета бионженерии и информатики МГУ им. Ломаносова рассказал ПостНауке о лаборатории, сепуркомьютерах, которые используются для обработки геномных данных, и проблемах, которые в связи с этим приходится решать.

В 2003 году был завершен проект «Геном человека», начатый в начале 1990-х, благодаря которому был секвенирован геном человека, хотя в нем и остается лакуны.
Ниже табличка, на которой показана динамика развития исследований:
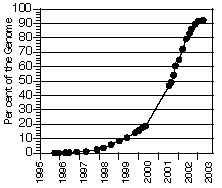
Чтобы секвенировать геном было потрачено 3 миллиарда рублей и огромное количество усилий ученых, собранных в международный проект. Сейчас ситуация изменилась с изобретением новых приборов высокопроизводительного секвенирования, и можно прочитать 10 геномов за 2 недели и за не высокую плату. Об этом уже рассказывал микробиолог Константин Северинов, приводя в пример европейский и американский опыт, когда ученые отдают на аутсорс сторонним фирмам разные практические и экспериментальные задачи.
Главная трудность, стоящая сейчас перед биоинформатиками – создание специального компьютера и его обслуживание. Для этого необходим специалист, который будет отвечать за его архитектуру. Таких людей, однако, в России совсем немного. Зачем он нужен: потому что если не выделить персонально ответственное лицо, то архитектура будет сформирована на основе уже имеющихся решений, которые могут не подходить для задачи обработки геномных данных.
Но когда есть архитектор и компьютер, то встают новые задачи:
1. Нужно решить, какую операционную систему использовать.
Скажем, в лаборатории эволюционной геномики факультета бионженерии и информатики МГУ им. Ломаносова стоит операционка Scientific Linux, разработанная на основе промышленного дистрибутирования Redhat Enterprise Linux ведущими ЦЕРНа и Фирмы Lab.
2. Проблема использования файловых систем. Опять же в МГУ используют файловые системы Lustre (развернутая файловая система, которая обычно используется в очень мощных суперкомпьютерах, в частности на Ломоносове, и позволяет распределить нагрузку на дисковые массивы) и OCFS2, XFS.
3. Задача мониторинга системы. В МГУ это Nagios. Такая система нужна, чтобы администратор сразу знал, если что-то сломалось или выключилось.
4. Проблема конфигурации узлов. Ее в МГУ решает система Puppet, которая позволяет автоматически настраивать конфигурацию всех узлов.
При этом все вышеописанное для специалистов является общим местом, а вот когда биологи сталкиваются с этой проблемой, то начинают ее решать с нуля. В России только начинается процесс оборудования лабораторий секвенаторами, и когда это случается, то возникает проблема оборудования для обработки данных секвенирования, потому что биологи, как признается Науменко, не умеют обращаться ни с чем мощнее ноутбука.
И от этого они вынуждены обращаться либо к вендорам за помощью, либо к физикам, которые используют подобные компьютеры со времен расчетов атомной водородной бомбы. Однако их компьютеры не подходят под задачи биологов.
Чем отличается суперкомпьютер от компьютера в лаборатории эволюционной геномики МГУ: Суперкомпьютер (их список можно найти на supercomputers.ru) – это мощный вычислитель с огромным количеством процессоров, очень быстрой сетью связей между процессорами и с относительно маленьким хранилищем данных. А для биологов нужен компьютер, который обрабатывает данные, и который может вместить огромное количество данных, передавать их с большой скоростью, но который при этом обладает сравнительно небольшой вычислительной мощностью. То есть в нем количество процессоров сравнимо с количеством хранилищ данных.
Такой компьютер и есть в лабе МГУ. Он содержит около 500 терабайт дисковых массивов, это примерно одна треть от дисковых носителей суперкомпьютера Ломоносов, и содержит около 300-400 вычислительных ядер.

Компьютер Ломоносов
Компьютер в лаборатории эволюционной геномики МГУ решает следующие задачи: получает данные с двух секвенаторов и на нем же производится сборка геномов de novo из коротких чтений, аннотация, то есть разметка их на области кодирующие белки и не кодирующие белки, и просто задачи обработки сырых данных, которые поступают с секвенаторов.
Чтобы решить эти задачи пришлось сделать узлы с большой памятью – для сборки геномов de novo нужен большой объем оперативной памяти — 512 гигабайт оперативной памяти.
Проблема, которая как ни странно стоит прямо, — это проблема электричества, даже в МГУ бывают перебои и надо устанавливать мощные источники бесперебойного питания, чтобы в критических случаях питать от 5 минут до получаса всю компьютерную систему.
С появлением высокопроизводительных секвенаторов впервые появилась возможность дешево получать геномные данные на уровне полного генома, а не каких-то участков определенных генов, и это открыло совершенно новые возможности в эволюционной и медицинской геномике. Можно взять популяцию и прочитать геномы сразу 20-50 образцов, и проверить всю популяционную генетику, просто исходя из генотипов этих организмов. В медицине для любых предсказаний нужно множество повторов, то есть нужно отсеквенировать 50-100 пациентов, чтобы можно было о чем-то говорить. Поэтому и нужны секвенаторы, и нужно обрабатывать такие огромные объемы данных.
Поэтому сейчас биологам приходится переучиваться, учиться писать программы, изучать статистику, чтобы понимать смысл данных.
«Может быть когда-то эта эпоха пройдет и сменится более разумным подходом, когда будут планировать эксперименты и понимать, какие данные нужно получать, какие не нужно, но сейчас идет время завоевания прерий, когда секвенируют все что можно и получают все данные, до которых могут дотянуться и пытаются их обработать. Поэтому в связи с этим потребность компьютеров будет возрастать, пока не будет выработана более зрелая методология исследований» — говорит Сергей Науменко.
Автор: JuliaP








