В предыдущих частях От белков к РНК, Мат. критерии, Как уменьшить число поворотов цепи?, Как оценить ход сворачивания односпиральной РНК?, Ограничение оптимизирующих методов в играх с противником и без, Одна фундаментальная проблема, Введение в сворачивание многоспиральных РНК я рассказал основы к предлагаемому мной кибернетико-геометрическому подходу для задачи сворачивания РНК. Повторю формулировку задачи:
Имеем произвольную, реально существующую, первичную последовательность до 100 нуклеотидов. Знаем все водородные связи которые нужно образовать. На выходе получаем файл .pdb, в котором третичная структура из указанной первичной последовательности и где образованы все требуемые водородные связи.
Здесь я расскажу о практике, чтобы каждый мог попробовать что это такое. Мной было разработано ПО для расчета того, о чем я рассказывал. Здесь я даю ссылку на демо версию. И объясняю, что вы сможете увидеть. Ведь лучше один раз увидеть, чем 100 раз услышать :)
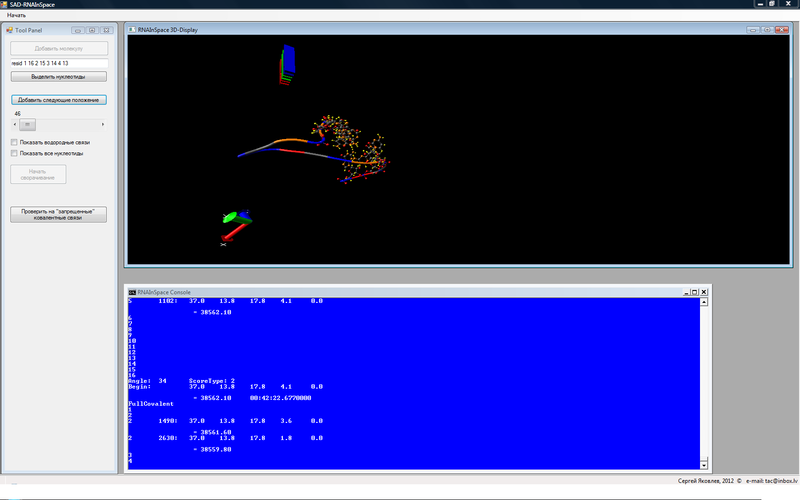
После того как Вы скачаете (качать от сюда), разархивируете, и запустите RNAInSpace.exe, нажмите «Начать» увидите экран (только с пустым верхним и нижним окном) (известен существующий баг, иногда верхнее окно может нормально не стать на свое место — просто перезапустите и повторите):
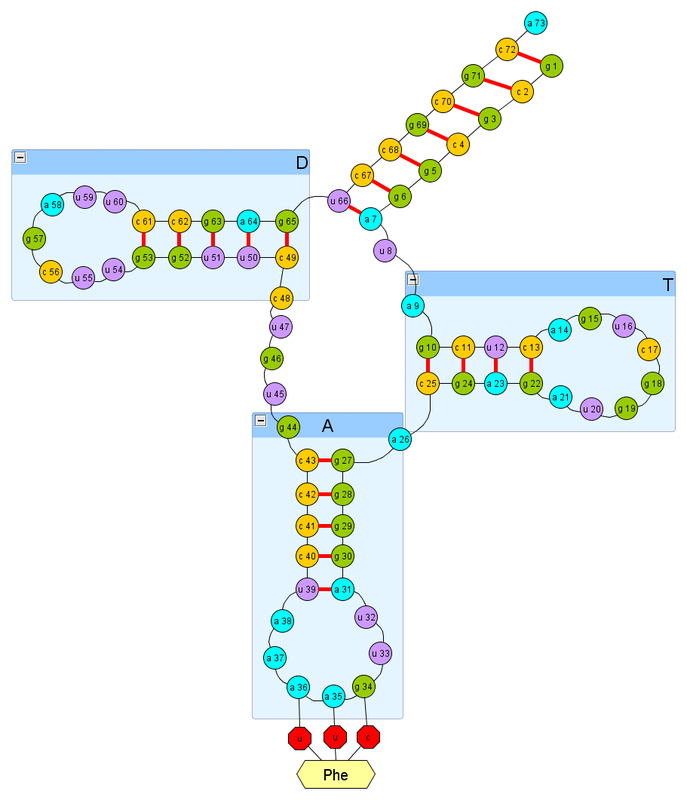
Это действительно демо-версия, вы не сможете управлять алгоритмом сворачивания. Но в остальном вы сможете проследить как происходит сворачивание. Изначально версия настроена на сворачивание одной спирали T-петли транспортной РНК организма Escherichia coli, которая переносит фенилаланин, ниже её вторичная структура (та часть, которая помечена «T»):
Теперь как управлять демо-версией.
Первое что вы делаете после загрузки — это нажимаете на кнопку «Начать сворачивание». В нижнем экране консоли начнут появляться цифры. О них позже. Проверьте в директории, где находится RNAInSpace.exe, начали появляться файлы X_#.pdb Как только появился X_0.pdb — нажимайте на кнопку «Добавить молекулу». На экране вверху Вы увидите цепь РНК вытянутую в линию. Можете нажать кнопку «Выделить нуклеотиды». Появятся 4 пары нуклеотидов, которые должны образовать между собой водородные связи (но их можно поменять, введя выше номера нуклеотидов, после слова resid, которые вы хотите отобразить). Два CheckBox'а ниже показывают/скрывают водородные связи (если они есть) и все нуклеотиды цепи.
Как только появляется следующий X_1.pdb, X_2.pdb, X_3.pdb и т.д. вы можете нажимать кнопку «Добавить следующие положение» и увидите как РНК начинает понемногу сворачиваться.
Графическое окно можно расширить. А также управляя мышкой можно вращать молекулу (жмем левую/правую кнопку и двигаем — вращается, крутим колесико увеличиваем/уменьшаем).
Если Вы выключите программу, и запустите заново, сворачивание начнется не сначала, а с последнего шага на котором остановилось сворачивание в предыдущий раз. Если вы захотите начать именно с начала, то нужно очистить файл traj.dat. И чтобы не запутаться, при следующем запуске, следует удалить все старые файлы X_#.pdb и X_#.txt. Формат .pdb вы также можете просматривать в стандартных ПО молекулярной визуализации, например, PyMOL.
Демо-версия не гарантирует, что спираль свернется — это как повезет :)
Как только наиграетесь с визуальным представлением и захотите разобраться, что значат цифры в консоле (впрочем если вы внимательно читали прошлые статьи, можно догадаться) во время расчетов и что еще можно сделать на демо-программе, или просто появятся вопросы спрашивайте, в следующей части постараюсь обобщить вопросы и сделать FAQ. Конечно, если потребуется и будет интерес.
P.S. Может быть кому-то не повезет, у меня на ноутбуке графика не загружается, пока не разбирался в чем проблема. Пишите о своих проблемах, по мере возможности буду смотреть.
upd. Я тут параллельно тестирую эту демо-программу. На 192 шаге (файл X_192.pdb) как раз появляется интересная ситуация, которую я описывал в Часть №5. Одна фундаментальная проблема. Там образуется водородные связи между концами спирали (1-16 нуклеотиды) в то время как внутри спирали еще не образованы. Дальше они будут рваться и образовываться вначале ближе к петле.
Автор: tac






