Рубрика «Фолдинг белков»
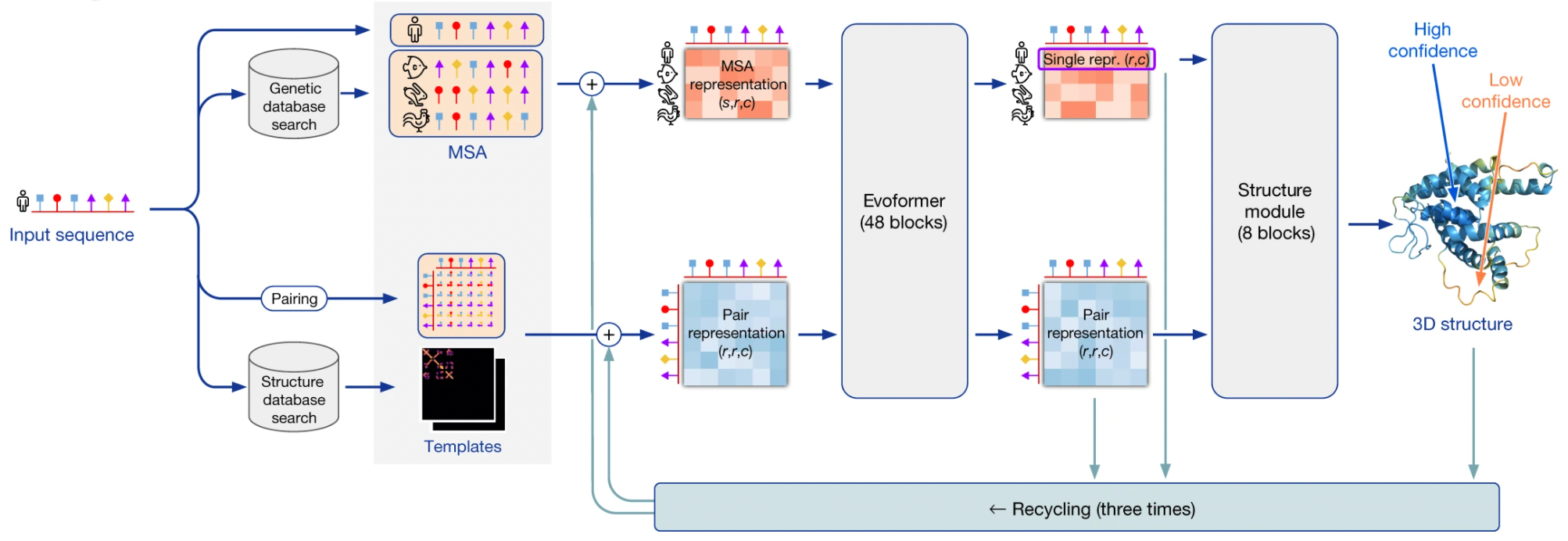
Как работает DeepMind AlphaFold2?
2022-01-08 в 18:50, admin, рубрики: AlphaFold2, DeepMind, биоинформатика, биология, Биотехнологии, машинное обучение, механизм внимания, нейронные сети, трансформеры, Фолдинг белковКак понять, что нейросеть решит вашу проблему. Прагматичное руководство
2020-06-19 в 7:00, admin, рубрики: Mail.Ru Cloud Solutions, nlp, PAC learning, Алгоритмы, Блог компании Mail.Ru Group, ВПК-обучение, детерминированные проблемы, искусственный интеллект, машинное обучение, нейросеть, обработка изображений, предикативное обслуживание, решение проблем, Фолдинг белков
Haystacks at Sunset Reimagined by AshnoAlice
Инженер по машинному обучению Джордж Хосу задает вопрос: «Какие проблемы решает машинное обучение?». Или конкретнее, с учетом современного развития отрасли: «Какие проблемы нейросеть способна решить на практике?». Команда Mail.ru Cloud Solutions перевела статью, так как рассуждения на эту тему, как нам кажется, встречаются редко.
Читать полностью »
Биовычисления по сворачиванию. Снова простым языком о полученной модели сворачивания
2012-06-15 в 23:33, admin, рубрики: Алгоритмы, биоинформатика, кибернетика, сворачивание рнк, теория игр, Фолдинг белков, метки: кибернетика, сворачивание рнк, теория игр, Фолдинг белковЯ тут написал уже более 7 статей на тему одного своего подхода (набора алгоритмов и проблем) к задаче сворачивания РНК. Читающих становилось с каждой статьей все меньше, а кое кто признавался, что мозг выносило уже после второй статьи. Сравнительный успех первых двух статей, по сравнению с остальными — кажется заключается в простоте изложения и не углубления в детали. Хотя последние статьи давали возможность самим взять демо моей программы и прочувствовать проблематику — это видимо интересует меньше.
Поэтому я постараюсь тут изложить простым языком еще одну проблему, которая мешает решить эту задачу. И мне представляется, что это проблема связанна не только с выбранным мной подходом к решению, а она скорее общая для задачи.
В своем ПО RNAInSpace — я реализовал возможность «покрутить» спираль РНК вручную, чтобы стала понятна геометрия и ограничения такого вращения. Но так как по предыдущим статьям — это ПО не сильно заинтересовало, то тут очередную демо версию этого ПО я представлять не буду. А поговорим о том, что получается у меня.
История одного реинжиниринга или RNAInSpace v.1.3. Demo
2012-05-16 в 3:30, admin, рубрики: биоинформатика, кибернетика, Проектирование и рефакторинг, реинжиниринг, сворачивание рнк, теория игр, Фолдинг белков, метки: demo, кибернетика, реинжиниринг, сворачивание рнк, теория игр, Фолдинг белковТут недавно был такой пост Правила разработки сложных систем. История одного проекта, где автор описывает как он удачно «копался» в одном проекте, а потом все выкинул и переписал с нуля.
Я попробую рассказать обратную историю. Тут около месяца назад я не удачно попытался представить демо версию одной своей разработки (см. Часть №7. RNAInSpace — программное обеспечение для полуавтоматического конструирования РНК в пространстве).
Оказалось, что у скачивающих не работает один модуль, ответственный за показ графики. В двух словах проект RNAInSpace — это программное обеспечение для полуавтоматического конструирования РНК в пространстве. Обеспечивает 3D визуализацию структуры РНК, позволяет её изменять и с помощью связи с модулем RNAWorld позволяет автоматизировать некоторые этапы сворачивания РНК.
Чтобы войти в тему — я тут написал некоторое множество статей:
От белков к РНК, Мат. критерии, Как уменьшить число поворотов цепи?, Как оценить ход сворачивания односпиральной РНК?, Ограничение оптимизирующих методов в играх с противником и без, Одна фундаментальная проблема, Введение в сворачивание многоспиральных РНК
Но эту статью можно обсуждать и не зная предметной области, кстати заодно проверим можно ли судить о качестве ПО не зная семантики предметной области (я утверждаю, что можно).
Так вот эта 3D визуализация (модуль RNAInSpaceDisplay) и не работала на некоторых компьютерах. Для реализации графики я использовал существующий проект VMD 1.8.7.
Ниже история о том как я адаптировал VMD 1.8.7 под свои нужды.
Часть №7. RNAInSpace — программное обеспечение для полуавтоматического конструирования РНК в пространстве
2012-03-30 в 13:34, admin, рубрики: биоинформатика, кибернетика, Программирование, разработка, сворачивание рнк, теория игр, Фолдинг белков, метки: demo, кибернетика, сворачивание рнк, теория игр, Фолдинг белковВ предыдущих частях От белков к РНК, Мат. критерии, Как уменьшить число поворотов цепи?, Как оценить ход сворачивания односпиральной РНК?, Ограничение оптимизирующих методов в играх с противником и без, Одна фундаментальная проблема, Введение в сворачивание многоспиральных РНК я рассказал основы к предлагаемому мной кибернетико-геометрическому подходу для задачи сворачивания РНК. Повторю формулировку задачи:
Имеем произвольную, реально существующую, первичную последовательность до 100 нуклеотидов. Знаем все водородные связи которые нужно образовать. На выходе получаем файл .pdb, в котором третичная структура из указанной первичной последовательности и где образованы все требуемые водородные связи.
Здесь я расскажу о практике, чтобы каждый мог попробовать что это такое. Мной было разработано ПО для расчета того, о чем я рассказывал. Здесь я даю ссылку на демо версию. И объясняю, что вы сможете увидеть. Ведь лучше один раз увидеть, чем 100 раз услышать :)
Часть №6. Введение в сворачивание многоспиральных РНК
2012-03-29 в 13:23, admin, рубрики: Алгоритмы, биоинформатика, Биотехнологии, кибернетика, математика, сворачивание рнк, теория игр, Фолдинг белков, метки: кибернетика, математика, сворачивание рнк, теория игр, Фолдинг белковИтак, в прошлых частях мы разобрались как сравнительно просто сворачивать спирали РНК. Теперь нам предстоит понять, как вообще сворачивается РНК. То РНК, которые мы взяли в виде примера имеет три спирали. Две из них L1 и L2 можно свернуть независимо. А вот с третьей проблемы. Эта третья состоит из концов РНК, и при ее сворачивании начинаю двигать наши свернутые спирали L1 и L2. Во-первых, при этом они мешают друг другу, и следовательно и сворачиванию третьей спирали. Во-вторых, возможно образование около десятка разнообразных псевдосимметричных структур — спирали L1, L2 могут по разному располагаться по отношению к сворачиваемым концам РНК.
Здесь мы попробуем разобраться как эти проблемы решить.
Часть №5. Биовычисления по сворачиванию. Одна фундаментальная проблема
2012-03-25 в 3:58, admin, рубрики: Алгоритмы, биоинформатика, Биотехнологии, кибернетика, математика, сворачивание рнк, теория игр, Фолдинг белков, метки: кибернетика, математика, сворачивание рнк, теория игр, Фолдинг белковВ этой статье мы рассмотрим как свернуть одну спираль в РНК. Для понимания нужно прочитать все предыдущие части От белков к РНК, Мат. критерии, Как уменьшить число поворотов цепи?, Как оценить ход сворачивания односпиральной РНК?, Ограничение оптимизирующих методов в играх с противником и без. Если ранее у нас все шло как по маслу, то здесь мы столкнемся с серьезной проблемой. Может кто-то подскажет как её решить.
Часть №4. Биовычисления по сворачиванию. Как оценить ход сворачивания односпиральной РНК?
2012-03-19 в 6:57, admin, рубрики: Алгоритмы, биоинформатика, Биотехнологии, кибернетика, математика, сворачивание рнк, теория игр, Фолдинг белков, метки: кибернетика, математика, сворачивание рнк, теория игр, Фолдинг белковИтак, если еще не устали от цикла «Hello, RNA World» — ловите последнею статью сезона :)
В прошлой статье я обосновал, почему следует (или хотя бы целесообразно) отказаться от оценки энергии как целевой функции. Если кто не в курсе — целевая функция, это такая придуманная нами функция, по которой можно оценить приближаемся мы к поставленной нами цели или нет, т.е. «правильно» сворачивается РНК или нет.
Если энергия — это мало репрезентативная цель, тогда что более стабильно/чётче указывает куда двигаться? Если бы у нас была абсолютно формализованная и точная цель — это уже означало бы, что мы задачу решили, т.к. сама формализация целевой функции — есть не что иное как полноценное понимание процесса.
Но у нас такой роскоши нет. Мы вынуждены вначале выдвигать гипотезу — каким закономерностям подчиняется процесс, и определенным образом отражать это в целевой функции.
Часть №3. Биовычисления по сворачиванию. Как уменьшить число поворотов цепи?
2012-03-17 в 22:23, admin, рубрики: Алгоритмы, биоинформатика, Биотехнологии, кибернетика, сворачивание рнк, теория игр, Фолдинг белков, метки: кибернетика, сворачивание рнк, теория игр, Фолдинг белковВ этой части мы поговорим о том, как можно так сократить число поворотов цепи, чтобы сократить рассчеты, но при этом не потеряв возможность попадания в нужные состояния.
Но вначале хочу обратиться к специалистам в этой области:
Вначале развею возможное недопонимание: я любитель, и не занимаюсь этой темой профессионально. Я заметил, что тут есть специалисты в этой теме. Странно, что я не читаю ваших статей, а вы читаете мои. Очень надеюсь, что эта ситуация поменяется. Я хочу почитать ваши статьи, и желательно написанные простым языком, и где вы даете ответы, а не отправляете в известном направлении в гугл. Просто у меня есть некий негативный опыт, когда только начинал ряд специалистов, которых удавалось находить в интернете делали умный вид и не помогали словом, и делом — а отправляли в указанном направлении. Здесь я пытаюсь рассказать свой маленький опыт — но может это позволит кому-то легче стартовать.
Тем же кто желает тут похоливарить. Давайте так. Я такой любитель — которому погоны специалистов значут мало, а наука такое дело требует повторяемости (а не бизнес-скрытности, это же не бизнес, чтобы скрывать детали своих алгоритмов и не публиковать их код?), поэтому просто разговоры меня интересуют мало. Но меня очень интересует когда мне показывают, что я занимаюсь немного не тем, и что есть люди которые действительно чего-то добились. Вот задача над которой я мучаюсь. Решите и покажите, что это просто — буду очень благодарен.
Я даю произвольную (реально существующую) первичную последовательность до 100 нуклеотидов. Указываю все водородные связи которые нужно образовать. Вы на выходе даете мне файл .pdb, в котором третичная структура из указанной первичной последовательности и где образованы все требуемые водородные связи. Ни каких других требований.
Прошу или показать, что это просто или ответственно подтвердить, что эта задача скажем за неделю (или другой разумный срок) — не решается.
Ну, а пока этого нет и нет ваших статей, например, о других подходах, вроде молекулярной динамики и т.д., извольте читать о предлагаемом мной подходе и критиковать конструктивно, помогать своими знаниями, участвовать в обсуждении проблемы и может быть даже объединить со мной усилия и чего-то сделать вместе.
И снова моей аудитории, которая не является специалистами: важно поверить, что это легко, и не обязательно знать физику, биологию, и сложную математику — надеюсь вы умеете программировать и этого достаточно. Выше кстати, задача, которую мы и будем решать… но не все сразу. По плюсам — я понял что Вы читаете. Но неужели все понятно и нет вопросов? Если что жду комментариев, даже самых наивных. Пора делать эту область исследований хотя бы простой по описанию, а не скрывать ее за не нужными тонами сложностей.
Часть №2. Введение в биовычисления по сворачиванию. Мат. критерии
2012-03-17 в 2:15, admin, рубрики: Алгоритмы, биоинформатика, Биотехнологии, кибернетика, сворачивание рнк, теория игр, Фолдинг белков, метки: кибернетика, сворачивание рнк, теория игр, Фолдинг белковЭто продолжение статьи Часть №1. Введение в биовычисления по сворачиванию. От белков к РНК. Здесь мы опишем ковалентные и водородные связи математически. Посмотрим какие углы мы будем вращать у РНК для сворачивания. И прикоснемся к вопросу «а в чем трудность то?»