Я тут написал уже более 7 статей на тему одного своего подхода (набора алгоритмов и проблем) к задаче сворачивания РНК. Читающих становилось с каждой статьей все меньше, а кое кто признавался, что выносило уже после второй статьи. Сравнительный успех первых двух статей, по сравнению с остальными — кажется заключается в простоте изложения и не углубления в детали. Хотя последние статьи давали возможность самим взять демо моей программы и прочувствовать проблематику — это видимо интересует меньше.
Поэтому я постараюсь тут изложить простым языком еще одну проблему, которая мешает решить эту задачу. И мне представляется, что это проблема связанна не только с выбранным мной подходом к решению, а она скорее общая для задачи.
В своем ПО RNAInSpace — я реализовал возможность «покрутить» спираль РНК вручную, чтобы стала понятна геометрия и ограничения такого вращения. Но так как по предыдущим статьям — это ПО не сильно заинтересовало, то тут очередную демо версию этого ПО я представлять не буду. А поговорим о том, что получается у меня.
Чтобы несколько поддержать разговор с моими читателями пройдусь по ряду их важных замечаний.
chupvl уже спрашивал Вы тестировали ваше решение на реальной задаче, или просто разбираете теорию на данный момент? Там я ему пояснил так:
Я сворачиваю рибозим. Рибозимы имеют одинаковую базовую структуру, точнее то из-за чего их выделяют в один класс. Для сравнения я использую другой реально имеющийся рибозим 2QUS. Мне потом важно увидеть в чем отличия, а в чем похожесть.
Конкретный пример сворачивания
А теперь хочу наглядно продемонстрировать, как выглядит та РНК которую я сворачиваю.
Ниже «скелет» РНК, который я использую как базовый (2QUS).

Я же пытаюсь свернуть вироидный рибозим NC_003540, третичная структура которого неизвестна. Но она должна быть в чем то подобна базовому. Хотя первичная последовательность у них почти на 80% разная. Ниже один из наилучших вариантов, которые мне удалось получить. (не автоматически, а полуавтоматически — аналогично как если бы играть в игру FoldIt только другим способом, я уже как то говорил, что до полной автоматизации — как до луны)

Какие отличия мы тут можем видеть?
1. Надо понимать, что базовая последовательность (2QUS) несколько длиннее, чем в вироидном рибозиме, поэтому концы РНК в вироидном рибозиме не будут образовывать длинную спираль, как в базовом аналоге.
2. Общая укладка принципиально совпадает — поэтому вироидный рибозим принципиально свернут верно.
3. Но есть как минимум одно отличие. Петля L2 (см. участок на рисунках слева снизу) — отличается.
3.а. В этой петле есть один нуклеотид, который должен образовать 4 водородных связи с двумя другими нуклеотидами в петле L1 (участок на рисунках справа снизу). Именно эти связи и удерживают петли рибозима вместе.
(как это наглядно выглядит и какие тут проблемы, которые я уже решил — я комментировал ранее здесь и здесь)
3.б. Почему же существует отличие? В базовом рибозиме эта петля просто длиннее, чем в изучаемом мной вироидном рибозиме, и петле L2 приходится менять свою конфигурацию, чтобы она могла состыковаться с петлей L1. А так как это достаточно неестественное положение петли L2, вместе с этим должна меняться конфигурация самой спирали (то, что выше слева).
4. И наконец, после разрешения всех этих проблем, я уже потирал руки, думая, что свернуть то концы рибозима при достаточно правильном положении ядра рибозима — не составит проблем. Но ошибся. Мне так и не удалось образовать нужные водородные связи между концами спирали (на рисунке можно видеть, что концы находятся не достаточно близко друг к другу).
Почему же не удалось досвернуть рибозим?
Я начал сравнивать, в чем же проблема у свернутой мной структуре РНК. Концы не хотели сходится, т.к. им мешал выступ соединяющий спирали L1 и L2 (по центру сверху). И оказалось, что буквально один-два нуклеотида, после этого выступа заняли не верное положение. Им в моделировании не очень много отводилось внимания. Они не образовывали никаких водородных связей — и я им позволил принимать любое возможное положение. И они приняли — случайное. И естественно, случайное положение не позволило досвернуться рибозиму. (ответвление: только подумайте — как может, что-то получится при моделировании, если в большинстве теперь используемых методах нуклеотиды как раз ставятся в случайное положение, а выбираются правильные по каким то среднестатистическим для всей молекулы РНК показателям?)
Я попробовал изменить это положение в уже свернутой структуре (второй рисунок). Но это оказалось не возможно — нужно перестраивать практически все связи, т.е. разрушать все те водородные связи которые уже были образованы.
И тут я задумался. Вспомнил комментарий от Wott: начальное состояние диктует результат, и ранее
И собственно начальное положение и диктует какое именно «конечное» состояние будет достигнуто. А начальное состояние в этом случае — сам процесс создания цепочки. То есть по хорошему задачу надо решать итерационно — добавили первый, добавили следующий — привели систему в минимум, добавили следующий — опять привели в минимум.
…
Точно также вредно рассматривать вытянутую цепочку целиком — это неправдоподобное состояние из которого можно достигнуть как правдоподобного, так и не очень состояний, но в силу большой величины вариантов, вторых будет существенно больше.
Это достаточно верное замечание, и для меня оно было известно, ранее в одной своей научной статье я писал:
…
В терминологии теории игр это означает, что нужно обеспечить возможность из любого начального положения игры достичь заданного конечного состояния. И если, например, в игре в шахматы мы знаем, как начинается игра и какое условие выигрыша, и знаем, что правилами шахмат обеспечено попадание из начала игры в конец, то здесь — при моделировании сворачивания макромолекул — нам нужно еще найти такие правила и доказать, что они обеспечивают процесс сворачивания.
Поэтому тогда я и ответил в целом, что такой подход противоречит моим результатам, и что лучше уж начинать сворачивать из начального состояния вытянутого в цепочку, чем из полусвернутого.
Но существуют еще так называемые стэкинг взаимодействия: это когда нуклеотиды, которые упрощенно можно назвать шестиугольниками расположены так, что образуют как бы стопку монет. Ниже на рисунке первые 6 нуклеотидов (считая снизу) находятся в стэкинг взаимодействии.
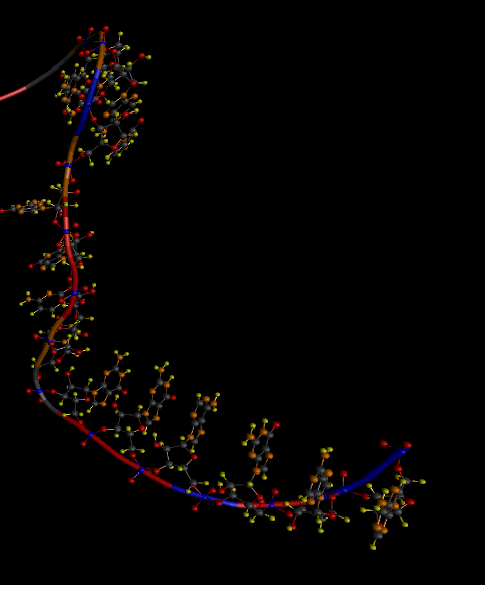
Так вот если РНК появляется постепенно, то до того как может образоваться первая водородная связь нужно чтобы появилось как минимум двадцать нуклеотидов.
И раньше я считал, что они просто вытянуты в цепь. Но они на самом деле наверняка появляясь один за другим успевают принять положение свойственное стэкингу. И цепочка вроде как вятянута, но уже не совсем случайно. И это вот как раз и важно для начального положения.
Но тут оказалась еще одна неприятность. Если мы вспомним как выглядит двойная цепь ДНК — то её длина может быть сильно большой. А там как раз именно такие стэкинг взаимодействия. Но РНК хотя и стремится к этому, но её хватает только на очень не большие участки, и тут на третьем рисунке вы видите как начиная с 7 нуклеотида цепь РНК меняет направление.
Некоторые выводы
Наверное не очень понятно к чему я вел все это время. Попробуем разобраться.
1. Стэкинг оказывается важен как создание начального положения, которое способствует тому, чтобы при сворачивании в природе не получилось ситуации как у меня, в описываемом выше моделировании, когда РНК практически свернулось — но пару нуклеотидов остались в случайном положении (не связанные стэкингом) — и это мешает дальшейшему сворачиванию.
2. Стэкинг РНК не такой стабильный как в ДНК, где образуется строгая спираль. У РНК в некоторых узловых точках прекращает между парой идущих друг за другом нуклеотидов действовать силы стэкинга. И тогда цепь меняет направление.
3. Там где меняется направление цепи, и тогда когда находятся комплементарные пары нуклеотидов — то вот тогда и начинаю создаваться водородные связи, которые имеют более сильное стабилизирующие действие, чем стэкинг.
4. Но когда цепь вырастает, и разделяется на две и больше спиралей — петли этих спиралей соединяются между собой нестандартными водородными связями. Это еще более сильное взаимодействие, и в это время может рушится предшествующий стэкинг, приближение водородных связей, и меняться форма начиная с петель спиралей.
Вот это та гипотеза — модель сворачивания, которая мной получена на данный момент. Её еще надо проверить окончательно, но сейчас проверены как минимум все другие вариации, которые не могут быть справедливыми (типа иерархической модели (где говорится вначале о создании вторичных структур, и только потом их совмещении в третичную) и прочих).
В итоге получается, что задача стэкинга подтолкнуть к образованию нужных водородных связей. А без его наличия водородные связи сами по себе не образуются.
P.S. На самом деле, все немного сложнее, но не хочу вас грузить. И так похоже обещал просто, а получилось не совсем. Но готов ответить на любые вопросы, где что-то не ясно.
Автор: tac






![Azure понятным языком [Шпаргалка] Azure понятным языком [Шпаргалка]](https://www.pvsm.ru/wp-content/plugins/contextual-related-posts/timthumb/timthumb.php?src=http%3A%2F%2Fwww.pvsm.ru%2Fimages%2F2017%2F01%2F31%2FAzure-ponyatnym-yazykom-shpargalka.jpg&w=100&h=100&zc=1&q=75)
