Что, если у вас идея для классного, полезного белка, и вы хотите получить его в реальности? Например, хотите создать вакцину против H. pylori (как словенская команда на iGEM 2008), создав гибридный белок, который сочетает фрагменты флагеллина E. coli, стимулирующие иммунный ответ с обычным флагеллином H. pylori?
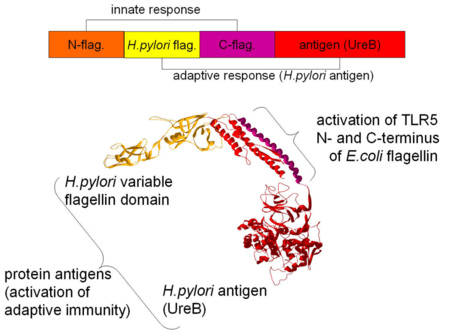
Дизайн гибридного флагеллина вакцины против H. pylori, представленный командой Словении на iGEM 2008
Удивительно, но мы очень близки к тому, чтобы создать любой белок, какой хотим, не выходя из блокнота Jupyter, благодаря последним разработкам в геномике, синтетической биологии и совсем недавно — в облачных лабораториях.
В этой статье я покажу код Python от идеи белка до его экспрессии в бактериальной клетке, не прикасаясь к пипетке и не разговаривая ни с одним человеком. Общая стоимость составит всего несколько сотен долларов! Используя терминологию Виджая Панде из A16Z, это Биология 2.0.
Конкретнее, в статье питоновский код облачной лаборатории делает следующее:
- Синтез последовательность ДНК, которая кодирует любой белок, который я хочу.
- Клонирование этой синтетической ДНК в вектор, который может её экспрессировать.
- Трансформация бактерии с этим вектором и подтверждение, что происходит экспрессия.
Настройка Python
Во-первых, общие настройки Python, которые нужны для любого блокнота Jupyter. Импортируем некоторые полезные модули Python и создаём некоторые служебные функции, в основном, для визуализации данных.
import re
import json
import logging
import requests
import itertools
import numpy as np
import seaborn as sns
import pandas as pd
import matplotlib as mpl
import matplotlib.pyplot as plt
from io import StringIO
from pprint import pprint
from Bio.Seq import Seq
from Bio.Alphabet import generic_dna
from IPython.display import display, Image, HTML, SVG
def uprint(astr): print(astr + "n" + "-"*len(astr))
def show_html(astr): return display(HTML('{}'.format(astr)))
def show_svg(astr, w=1000, h=1000):
SVG_HEAD = '''<?xml version="1.0" standalone="no"?><!DOCTYPE svg PUBLIC "-//W3C//DTD SVG 1.1//EN" "http://www.w3.org/Graphics/SVG/1.1/DTD/svg11.dtd">'''
SVG_START = '''<svg viewBox="0 0 {w:} {h:}" version="1.1" xmlns="http://www.w3.org/2000/svg" xmlns:xlink= "http://www.w3.org/1999/xlink">'''
return display(SVG(SVG_HEAD + SVG_START.format(w=w, h=h) + astr + '</svg>'))
def table_print(rows, header=True):
html = ["<table>"]
html_row = "</td><td>".join(k for k in rows[0])
html.append("<tr style='font-weight:{}'><td>{}</td></tr>".format('bold' if header is True else 'normal', html_row))
for row in rows[1:]:
html_row = "</td><td>".join(row)
html.append("<tr style='font-family:monospace;'><td>{:}</td></tr>".format(html_row))
html.append("</table>")
show_html(''.join(html))
def clean_seq(dna):
dna = re.sub("s","",dna)
assert all(nt in "ACGTN" for nt in dna)
return Seq(dna, generic_dna)
def clean_aas(aas):
aas = re.sub("s","",aas)
assert all(aa in "ACDEFGHIKLMNPQRSTVWY*" for aa in aas)
return aas
def Images(images, header=None, width="100%"): # to match Image syntax
if type(width)==type(1): width = "{}px".format(width)
html = ["<table style='width:{}'><tr>".format(width)]
if header is not None:
html += ["<th>{}</th>".format(h) for h in header] + ["</tr><tr>"]
for image in images:
html.append("<td><img src='{}' /></td>".format(image))
html.append("</tr></table>")
show_html(''.join(html))
def new_section(title, color="#66aa33", padding="120px"):
style = "text-align:center;background:{};padding:{} 10px {} 10px;".format(color,padding,padding)
style += "color:#ffffff;font-size:2.55em;line-height:1.2em;"
return HTML('<div style="{}">{}</div>'.format(style, title))
# Show or hide text
HTML("""
<style>
.section { display:flex;align-items:center;justify-content:center;width:100%; height:400px; background:#6a3;color:#eee;font-size:275%; }
.showhide_label { display:block; cursor:pointer; }
.showhide { position: absolute; left: -999em; }
.showhide + div { display: none; }
.showhide:checked + div { display: block; }
.shown_or_hidden { font-size:85%; }
</style>
""")
# Plotting style
plt.rc("axes", titlesize=20, labelsize=15, linewidth=.25, edgecolor='#444444')
sns.set_context("notebook", font_scale=1.2, rc={})
%matplotlib inline
%config InlineBackend.figure_format = 'retina' # or 'svg'Облачные лаборатории
Как у AWS или любого вычислительного облака, у облачной лаборатории есть оборудование для молекулярной биологии, а также роботы, которых она сдаёт в аренду через интернет. Вы можете выдавать инструкции своим роботам, нажав несколько кнопок в интерфейсе или написав код, который самостоятельно их программирует. Необязательно писать собственные протоколы, как я сделаю здесь, значительная часть молекулярной биологии — это стандартные рутинные задачи, поэтому обычно лучше полагаться на надёжный чужой протокол, который показал хорошее взаимодействие с роботами.
В последнее время появился целый ряд компаний с облачными лабораториями: Transcriptic, Autodesk Wet Lab Accelerator (бета, и построена на основе Transcriptic), Arcturus BioCloud (бета), Emerald Cloud Lab (бета), Synthego (ещё не запустилась). Есть даже компании, построенные поверх облачных лабораторий, такие как Desktop Genetics, которая специализируется на CRISPR. Начинают появляться научные статьи об использовании облачных лабораторий в реальной науке.
На момент написания статьи в открытом доступе только Transcriptic, поэтому будем использовать её. Насколько я понимаю, большая часть бизнеса Transcriptic построена на автоматизации общих протоколов, а написание собственных протоколов на Python (как я буду делать в этой статье) менее распространено.

«Рабочая ячейка» Transcriptic с холодильниками внизу и различным лабораторным оборудованием на стенде
Я выдам роботам Transcriptic инструкции на автопротоколе. Autoprotocol — это язык на основе JSON для написания протоколов для лабораторных роботов (и людей, как бы). Autoprotocol в основном сделан на этой библиотеке Python. Язык был первоначально создан и до сих пор поддерживается Transcriptic, но, как я понимаю, он полностью открыт. Есть хорошая документация.
Интересная идея, что на автопротоколе можно писать инструкции для людей в удалённых лабораториях — скажем, в Китае или Индии — и потенциально получить некоторые преимущества от использования и людей (их суждение), и роботов (отсутствие суждения). Здесь нужно упомянуть protocols.io, это попытка стандартизировать протоколы для повышения воспроизводимости, но для людей, а не роботов.
"instructions": [
{
"to": [
{
"well": "water/0",
"volume": "500.0:microliter"
}
],
"op": "provision",
"resource_id": "rs17gmh5wafm5p"
},
...
]Пример фрагмента autoprotocol
Настройки Python для молекулярной биологии
Кроме импорта стандартных библиотек, мне понадобятся некоторые специфические молекулярно-биологические утилиты. Этот код в основном для автопротокола и Transcriptic.
В коде часто встречается концепция «мёртвого объёма» (dead volume). Это означает последнюю каплю жидкости, который роботы Transcriptic не могут взять пипеткой из пробирок (потому что они не видят!). Приходится тратить много времени, чтобы убедиться, что в колбах достаточный объём материала.
import autoprotocol
from autoprotocol import Unit
from autoprotocol.container import Container
from autoprotocol.protocol import Protocol
from autoprotocol.protocol import Ref # "Link a ref name (string) to a Container instance."
import requests
import logging
# Transcriptic authorization
org_name = 'hgbrian'
tsc_headers = {k:v for k,v in json.load(open("auth.json")).items() if k in ["X_User_Email","X_User_Token"]}
# Transcriptic-specific dead volumes
_dead_volume = [("96-pcr",3), ("96-flat",25), ("96-flat-uv",25), ("96-deep",15),
("384-pcr",2), ("384-flat",5), ("384-echo",15),
("micro-1.5",15), ("micro-2.0",15)]
dead_volume = {k:Unit(v,"microliter") for k,v in _dead_volume}
def init_inventory_well(well, headers=tsc_headers, org_name=org_name):
"""Initialize well (set volume etc) for Transcriptic"""
def _container_url(container_id):
return 'https://secure.transcriptic.com/{}/samples/{}.json'.format(org_name, container_id)
response = requests.get(_container_url(well.container.id), headers=headers)
response.raise_for_status()
container = response.json()
well_data = container['aliquots'][well.index]
well.name = "{}/{}".format(container["label"], well_data['name']) if well_data['name'] is not None else container["label"]
well.properties = well_data['properties']
well.volume = Unit(well_data['volume_ul'], 'microliter')
if 'ERROR' in well.properties:
raise ValueError("Well {} has ERROR property: {}".format(well, well.properties["ERROR"]))
if well.volume < Unit(20, "microliter"):
logging.warn("Low volume for well {} : {}".format(well.name, well.volume))
return True
def touchdown(fromC, toC, durations, stepsize=2, meltC=98, extC=72):
"""Touchdown PCR protocol generator"""
assert 0 < stepsize < toC < fromC
def td(temp, dur): return {"temperature":"{:2g}:celsius".format(temp), "duration":"{:d}:second".format(dur)}
return [{"cycles": 1, "steps": [td(meltC, durations[0]), td(C, durations[1]), td(extC, durations[2])]}
for C in np.arange(fromC, toC-stepsize, -stepsize)]
def convert_ug_to_pmol(ug_dsDNA, num_nts):
"""Convert ug dsDNA to pmol"""
return float(ug_dsDNA)/num_nts * (1e6 / 660.0)
def expid(val):
"""Generate a unique ID per experiment"""
return "{}_{}".format(experiment_name, val)
def µl(microliters):
"""Unicode function name for creating microliter volumes"""
return Unit(microliters,"microliter")Синтез ДНК и синтетическая биология
Несмотря на связь с современной синтетической биологией, синтез ДНК — довольно старая технология. Мы в течение десятилетий умели делать оликонуклеотиды (то есть последовательности ДНК до 200 оснований). Однако это всегда было дорого, и химия никогда не допускала длинных последовательностей ДНК. В последнее время стало возможным по разумной цене синтезировать целые гены (до тысяч оснований). Это достижение действительно открывает эпоху «синтетической биологии».
Компания Synthetic Genomics Крейга Вентера продвинула синтетическую биологию дальше всего, синтезировав целый организм — более миллиона оснований в длину. По мере увеличения длины ДНК проблемой становится уже не синтез, а сборка (т. е. сшивание вместе синтезированных последовательностей ДНК). При каждой сборке вы можете увеличить длину ДНК в два раза (или больше), поэтому после десятка или около того итераций получается довольно длинная молекула! Различие между синтезом и сборкой довольно скоро должно стать понятным конечному пользователю.
Закон Мура?
Цена синтеза ДНК падает довольно быстро, с более $0,30 за основание два года назад до около $0,10 сегодня, но она развивается больше как бактерии, чем процессоры. Напротив, цены на секвенирование ДНК падают быстрее, чем закон Мура. Цель $0,02 за основание намечена как точка перегиба, где вы можете заменить много трудоёмких манипуляций с ДНК простым синтезом. Например, при такой цене вы можете синтезировать целую плазмиду 3kb за $60 и пропустить кучу молекулярной биологии. Надеюсь, мы достигнем этого через пару лет.

Цены на синтез ДНК по сравнению с ценами на секвенирование ДНК, цена за 1 основание (Carlson, 2014)
Компании по синтезу ДНК
В области синтеза ДНК есть несколько крупных компаний: IDT является крупнейшим производителем оликонуклеотидов, а также может производить более длинные (до 2kb) «фрагменты генов» (gBlocks). Gen9, Twist и DNA 2.0 обычно специализируются на более длинных последовательностях ДНК — это компании по синтезу генов. Есть также некоторые интересные новые компании, такие как Cambrian Genomics и Genesis DNA, которые работают над методами синтеза следующего поколения.
Другие компании, такие как Amyris, Zymergen и Ginkgo Bioworks, используют синтезированную этими компаниями ДНК для работы на уровне организма. Synthetic Genomics тоже делает это, но сама синтезирует ДНК.
Недавно Ginkgo заключила сделку с Twist на изготовление 100 миллионов оснований: самая крупная сделка, что я видел. Это доказывает, что мы живём в будущем, Twist даже рекламировала промокод в Twitter: когда вы покупаете 10 миллионов оснований ДНК (почти весь геном дрожжей!), вы получаете ещё 10 миллионов бесплатно.

Нишевое предложение Twist в твиттере
Часть первая: проектирование эксперимента
Зелёный флуоресцентный белок
В этом эксперименте синтезируем последовательность ДНК для простого, зелёного флуоресцентного белка (GFP). Протеин GFP впервые был найден в медузе, которая флуоресцирует под ультрафиолетовым светом. Это чрезвычайно полезный белок, потому что легко обнаружить его экспрессию, просто измеряя флуоресценцию. Существуют варианты GFP, которые производят жёлтый, красный, оранжевый и другие цвета.
Интересно посмотреть, как различные мутации влияют на цвет белка, и это потенциально интересная проблема машинного обучения. Совсем недавно для этого пришлось бы потратить немало времени в лаборатории, но сейчас я вам покажу, что это (почти) так же просто, как редактирование текстового файла!
Технически, мой GFP является суперфолдер-вариантом (sfGFP) с некоторыми мутациями для улучшения качеств.

В суперфолдер-GFP (sfGFP) некоторые мутации придают ему определённые полезные свойства
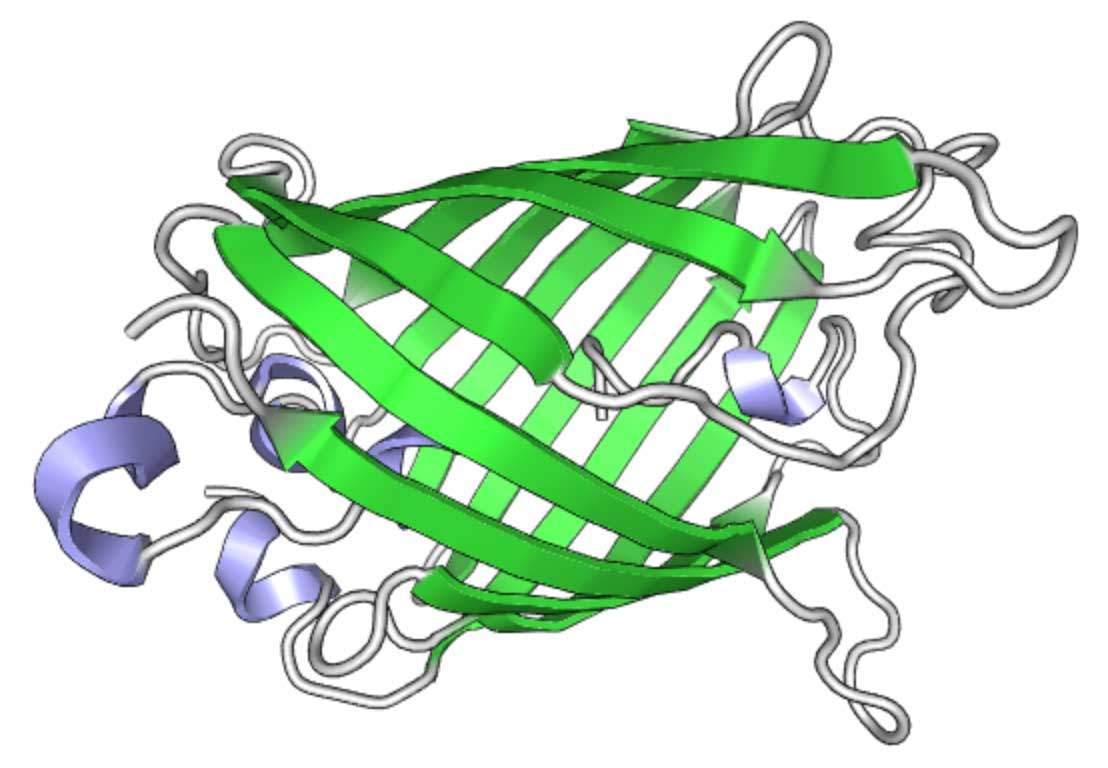
Структура GFP (визуализируется с помощью PV)
Синтез GFP в Twist
Мне посчастливилось попасть в альфа-программу тестирования Twist, поэтому я использовал их сервис для синтеза ДНК (они любезно разместили мой крошечный заказ — спасибо, Twist!). Это новая компания в нашей области, с новым упрощённым процессом синтеза. Цены у них в районе $0,10 за основание или ниже, но они всё ещё в бета-версии, а альфа-программа, в которой я принимал участие, закрылась. Twist поднял около $150 млн, так что их технология вызывает живой энтузиазм.
Я отправил свою последовательность ДНК в Twist в виде электронной таблицы Excel (пока нет API, но я предполагаю, что скоро будет), а они отправили синтезированную ДНК прямо на мой ящик в лаборатории Transcriptic (я также использовал для синтеза IDT, но они не отправили ДНК прямо в Transcriptic, что немного портит удовольствие).
Очевидно, этот процесс ещё не стал типичным случаем использования и требует некоторой поддержки, но он сработал, так что весь конвейер остаётся виртуальным. Без этого мне, вероятно, понадобился бы доступ в лабораторию — многие компании не будут отправлять ДНК или реагенты на домашний адрес.
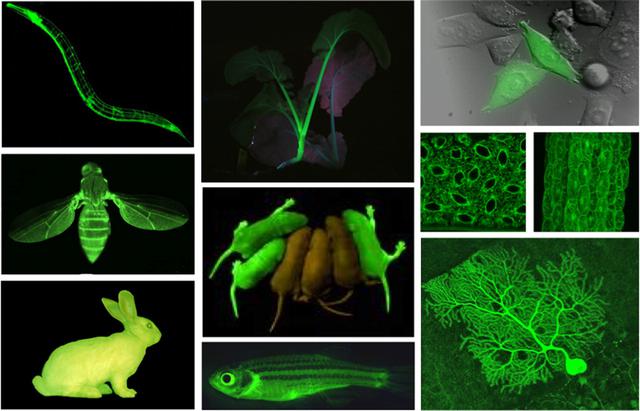
GFP безвреден, поэтому подсвечивается любой вид
Плазмидный вектор
Чтобы экспрессировать этот белок в бактериях, гену нужно где-то жить, иначе синтетическая ДНК, кодирующая ген, просто мгновенно деградирует. Как правило, в молекулярной биологии мы используем плазмиду, кусочек круглой ДНК, который живёт вне бактериального генома и экспрессирует белки. Плазмиды — удобный способ для бактерий делиться полезными автономными функциональными модулями, такими как устойчивость к антибиотикам. В клетке могут быть сотни плазмид.
Широко используемая терминология заключается в том, что плазмида является вектором, а синтетическая ДНК — инсерцией (вставкой). Итак, здесь мы пытаемся клонировать инсерцию в вектор, а затем трансформировать бактерии с помощью вектора.
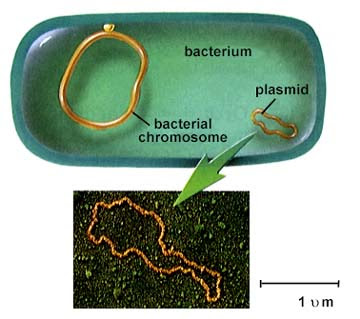
Бактериальный геном и плазмида (не в масштабе!) (Википедия)
pUC19
Я выбрал довольно стандартную плазмиду в серии pUC19. Эта плазмида очень часто используется, и поскольку она доступна как часть стандартного инвентаря Transcriptic, нам не нужно ничего им отправлять.
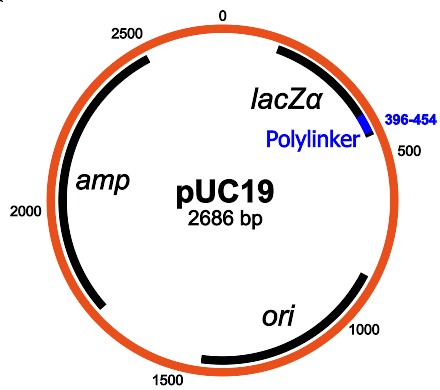
Структура pUC19: основными компонентами являются ген устойчивости к ампициллину, lacZα, MCS/полилинкер и происхождение репликации (Википедия)
У pUC19 есть приятная функция: поскольку он содержит ген lacZα, его можно использовать на сине-белом экране и посмотреть, в каких колониях успешно прошла инсерция. Нужны два химиката: IPTG и X-gal, и схема работает следующим образом:
- IPTG индуцирует экспрессию lacZα.
- Если lacZα деактивирован через ДНК, вставленную в место множественного клонирования (MCS/полилинкер) в lacZα, то плазмида не может осуществлять гидролиз X-gal, и эти колонии будут белыми вместо синих.
- Поэтому успешная инсерция производит белые колонии, а неудачная инсерция производит синие колонии.
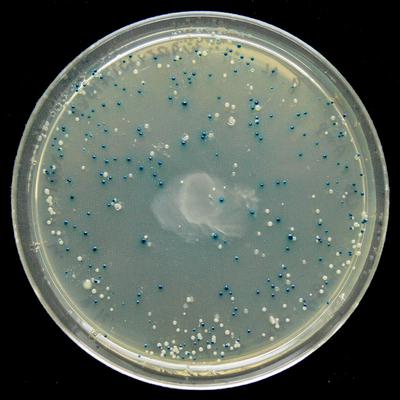
Сине-белый экран показывает, где была деактивирована экспрессия lacZα (Википедия)
Документация по openwetware говорит:
DH5α E. coli не требует IPTG, чтобы вызвать экспрессию от промотора lac, даже если в штамме экспрессируется репрессор Lac. Число копий большинства плазмид превышает число репрессоров в клетках. Если вам нужен максимальный уровень экспрессии, добавьте IPTG к конечной концентрации 1 mM.
Синтетические последовательности ДНК
Последовательность ДНК sfGFP
Легко получить последовательность ДНК для sfGFP, взяв последовательность белка и кодируя её кодонами, подходящими для организма-хозяина (здесь, E. coli). Это белок среднего размера с 236 аминокислотами, так что при 10 центах за основание синтез ДНК стоит около $70.
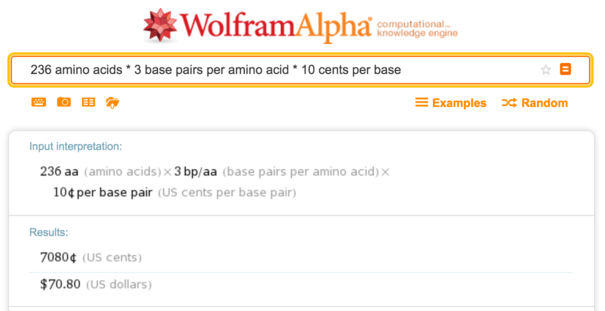
Wolfram Alpha, расчёт стоимости синтеза
Первые 12 оснований нашего sfGFP — это последовательность Шайна — Дельгарно, которую я добавил сам, что теоретически должно увеличить экспрессию (AGGAGGACAGCT, затем ATG (старт-кодон) запускает белок). Согласно вычислительному инструменту, разработанному Salis Lab (слайды лекции), мы можем ожидать от средней до высокой экспрессии нашего белка (скорость инициации перевода 10 000 «произвольных единиц»).
sfGFP_plus_SD = clean_seq("""
AGGAGGACAGCTATGTCGAAAGGAGAAGAACTGTTTACCGGTGTGGTTCCGATTCTGGTAGAACTGGA
TGGGGACGTGAACGGCCATAAATTTAGCGTCCGTGGTGAGGGTGAAGGGGATGCCACAAATGGCAAAC
TTACCCTTAAATTCATTTGCACTACCGGCAAGCTGCCGGTCCCTTGGCCGACCTTGGTCACCACACTG
ACGTACGGGGTTCAGTGTTTTTCGCGTTATCCAGATCACATGAAACGCCATGACTTCTTCAAAAGCGC
CATGCCCGAGGGCTATGTGCAGGAACGTACGATTAGCTTTAAAGATGACGGGACCTACAAAACCCGGG
CAGAAGTGAAATTCGAGGGTGATACCCTGGTTAATCGCATTGAACTGAAGGGTATTGATTTCAAGGAA
GATGGTAACATTCTCGGTCACAAATTAGAATACAACTTTAACAGTCATAACGTTTATATCACCGCCGA
CAAACAGAAAAACGGTATCAAGGCGAATTTCAAAATCCGGCACAACGTGGAGGACGGGAGTGTACAAC
TGGCCGACCATTACCAGCAGAACACACCGATCGGCGACGGCCCGGTGCTGCTCCCGGATAATCACTAT
TTAAGCACCCAGTCAGTGCTGAGCAAAGATCCGAACGAAAAACGTGACCATATGGTGCTGCTGGAGTT
CGTGACCGCCGCGGGCATTACCCATGGAATGGATGAACTGTATAAA""")
print("Read in sfGFP plus Shine-Dalgarno: {} bases long".format(len(sfGFP_plus_SD)))
sfGFP_aas = clean_aas("""MSKGEELFTGVVPILVELDGDVNGHKFSVRGEGEGDATNGKLTLKFICTTGKLPVPWPTLVTTLTYG
VQCFSRYPDHMKRHDFFKSAMPEGYVQERTISFKDDGTYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNFNSHNVYITADKQKN
GIKANFKIRHNVEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSVLSKDPNEKRDHMVLLEFVTAAGITHGMDELYK""")
assert sfGFP_plus_SD[12:].translate() == sfGFP_aas
print("Translation matches protein with accession 532528641")Read in sfGFP plus Shine-Dalgarno: 726 bases long Translation matches protein with accession 532528641
Последовательность ДНК pUC19
Сначала я проверяю, что у последовательности pUC19, которую я загрузил из NEB, правильная длина и она включает в себя ожидаемый полилинкер.
pUC19_fasta = !cat puc19fsa.txt
pUC19_fwd = clean_seq(''.join(pUC19_fasta[1:]))
pUC19_rev = pUC19_fwd.reverse_complement()
assert all(nt in "ACGT" for nt in pUC19_fwd)
assert len(pUC19_fwd) == 2686
pUC19_MCS = clean_seq("GAATTCGAGCTCGGTACCCGGGGATCCTCTAGAGTCGACCTGCAGGCATGCAAGCTT")
print("Read in pUC19: {} bases long".format(len(pUC19_fwd)))
assert pUC19_MCS in pUC19_fwd
print("Found MCS/polylinker")Read in pUC19: 2686 bases long Found MCS/polylinker
Делаем некоторые основные QC, чтобы убедиться, что EcoRI и BamHI присутствуют в pUC19 только один раз (следующие ограничительные ферменты имеются в инвентаре Transcriptic по умолчанию: PstI, PvuII, EcoRI, BamHI, BbsI, BsmBI).
REs = {"EcoRI":"GAATTC", "BamHI":"GGATTC"}
for rename, res in REs.items():
assert (pUC19_fwd.find(res) == pUC19_fwd.rfind(res) and
pUC19_rev.find(res) == pUC19_rev.rfind(res))
assert (pUC19_fwd.find(res) == -1 or pUC19_rev.find(res) == -1 or
pUC19_fwd.find(res) == len(pUC19_fwd) - pUC19_rev.find(res) - len(res))
print("Asserted restriction enzyme sites present only once: {}".format(REs.keys()))Теперь смотрим на последовательность lacZα и проверяем, что нет ничего неожиданного. Например, она должна начинаться с Met и заканчиваться стоп-кодоном. Также легко подтвердить, что это полный 324bp lacZα ORF, загрузив последовательность pUC19 в бесплатный инструмент просмотра snapgene.
lacZ = pUC19_rev[2217:2541]
print("lacZα sequence:t{}".format(lacZ))
print("r_MCS sequence:t{}".format(pUC19_MCS.reverse_complement()))
lacZ_p = lacZ.translate()
assert lacZ_p[0] == "M" and not "*" in lacZ_p[:-1] and lacZ_p[-1] == "*"
assert pUC19_MCS.reverse_complement() in lacZ
assert pUC19_MCS.reverse_complement() == pUC19_rev[2234:2291]
print("Found MCS once in lacZ sequence")lacZ sequence: ATGACCATGATTACGCCAAGCTTGCATGCCTGCAGGTCGACTCTAGAGGATCCCCGGGTACCGAGCTCGAATTCACTGGCCGTCGTTTTACAACGTCGTGACTGGGAAAACCCTGGCGTTACCCAACTTAATCGCCTTGCAGCACATCCCCCTTTCGCCAGCTGGCGTAATAGCGAAGAGGCCCGCACCGATCGCCCTTCCCAACAGTTGCGCAGCCTGAATGGCGAATGGCGCCTGATGCGGTATTTTCTCCTTACGCATCTGTGCGGTATTTCACACCGCATATGGTGCACTCTCAGTACAATCTGCTCTGATGCCGCATAG r_MCS sequence: AAGCTTGCATGCCTGCAGGTCGACTCTAGAGGATCCCCGGGTACCGAGCTCGAATTC Found MCS once in lacZ sequence
Сборка методом Гибсона
Сборка ДНК означает просто сшивание фрагментов. Обычно вы собираете несколько фрагментов ДНК в более длинный сегмент, а затем клонируете его в плазмиду или геном. В этом эксперименте я хочу клонировать один сегмент ДНК в плазмиду pUC19 ниже промотора lac для экспрессии в E. coli.
Существует множество способов клонирования (например, NEB, openwetware, addgene). Здесь я буду использовать сборку методом Гибсона (разработанную Даниэлем Гибсоном в Synthetic Genomics в 2009 году), которая не обязательно является самым дешёвым методом, зато простая и гибкая. Нужно только поместить ДНК, которую вы хотите собрать (с соответствующими перекрытиями) в пробирку со смесью Gibson Assembly Master Mix, и она собирается сама!
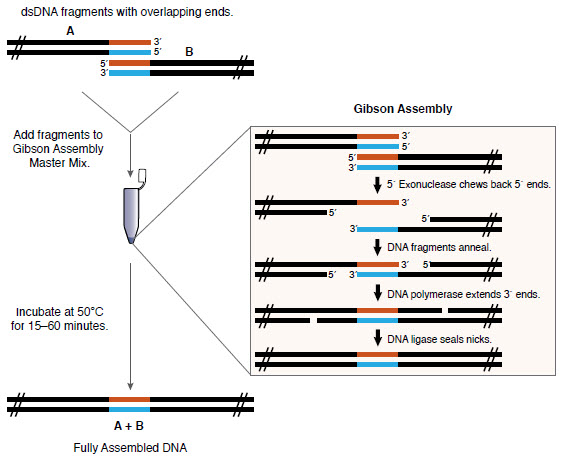
Обзор сборки Гибсона (NEB)
Исходный материал
Начинаем со 100 нг синтетической ДНК в 10 мкл жидкости. Это равняется 0,21 пикомолей ДНК или концентрации 10 нг/мкл.
pmol_sfgfp = convert_ug_to_pmol(0.1, len(sfGFP_plus_SD))
print("Insert: 100ng of DNA of length {:4d} equals {:.2f} pmol".format(len(sfGFP_plus_SD), pmol_sfgfp))Insert: 100ng of DNA of length 726 equals 0.21 pmol
Согласно протоколу сборки NEB, это достаточно исходного материала:
NEB рекомендует в общей сложности 0,02-0,5 пикомолей фрагментов ДНК, когда в вектор собирается 1 или 2 фрагмента, или 0,2-1,0 пикомолей фрагментов ДНК, когда собираются 4-6 фрагментов.
0.02-0.5 пмолей * X мкл
* Оптимизированная эффективность клонирования составляет 50-100 нг векторов с 2-3-кратным избытком инсерций. Используйте в 5 раз больше инсерций, если размер меньше 200 bps. Общий объём нефильтрованных фрагментов PCR в реакции сборки Гибсона не должен превышать 20%.
NEBuilder для сборки Гибсона
NEBuilder от компании Biolab — это действительно отличный инструмент для создания протокола сборки Гибсона. Он даже генерирует вам всеобъемлющий четырёхстраничный PDF со всей информацией. С помощью этого инструмента разрабатываем протокол для вырезания pUC19 с EcoRI, а затем используем PCR [ПЦР, полимеразная цепная реакция позволяет добиться значительного увеличения малых концентраций определённых фрагментов ДНК в биологическом материале — прим. пер.] для добавления фрагментов соответствующего размера в инсерцию.
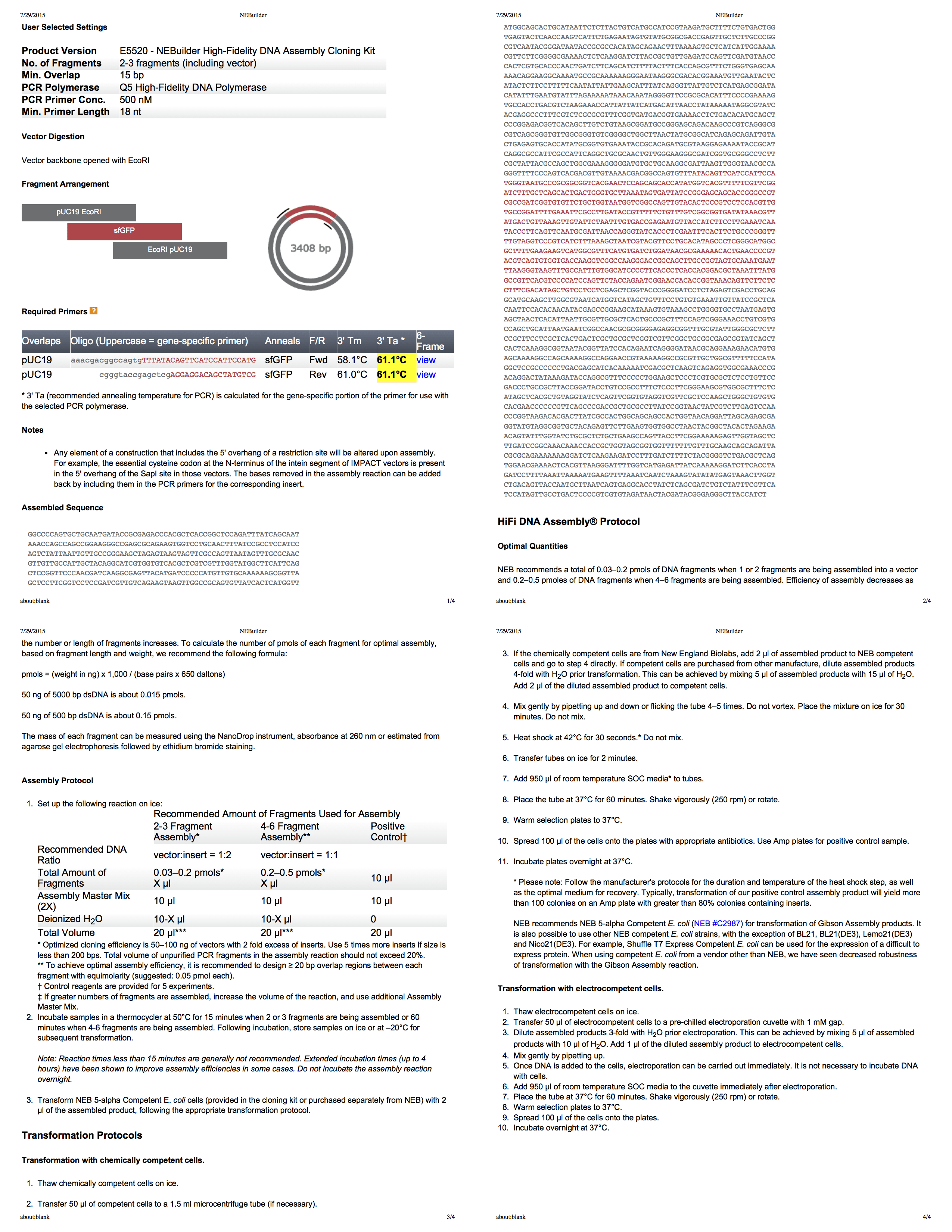
Часть вторая: проведение эксперимента
Эксперимент состоит из четырёх этапов:
- Полимеразная цепная реакция инсерции для добавления материала с фланкирующей последовательностью.
- Резка плазмиды для размещения инсерции.
- Сборка методом Гибсона инсерции и плазмиды.
- Трансформация бактерий с помощью собранной плазмиды.
Шаг 1. PCR инсерции
Сборка Гибсона зависит от последовательности ДНК, которую вы собираете, имея некоторую перекрывающуюся последовательность (см. выше протокол NEB с подробными инструкциями). Кроме простой амплификации, PCR также позволяет добавлять фланкирующую последовательность ДНК, просто включив дополнительную последовательность в праймеры (также можно клонировать, используя только OE-PCR).
Синтезируем праймеры в соответствии с протоколом NEB выше. Я попробовал протокол Quickstart на сайте Transcriptic, но есть ещё команда автопротокола. Transcriptic сама не производит синтез олигонуклеотидов, поэтому после 1-2 дней ожидания эти праймеры волшебным образом появляются в моём инвентаре (обратите внимание, что ген-специфическая часть праймеров ниже указана в верхнем регистре, но это просто косметические вещи).
insert_primers = ["aaacgacggccagtgTTTATACAGTTCATCCATTCCATG", "cgggtaccgagctcgAGGAGGACAGCTATGTCG"]Анализ праймеров
Можно проанализировать свойства этих праймеров с помощью IDT OligoAnalyzer. при отладке эксперимента PCR полезно знать температуры плавления и вероятность побочного эффекта primer dimer, хотя протокол NEB почти наверняка выберет праймеры с хорошими свойствами.
Gene-specific portion of flank (uppercase) Melt temperature: 51C, 53.5C Full sequence Melt temperature: 64.5C, 68.5C Hairpin: -.4dG, -5dG Self-dimer: -9dG, -16dG Heterodimer: -6dG
Я прошёл через много итераций PCR, прежде чем получить удовлетворительные результаты, включая эксперименты с несколькими различными марками смесей PCR. Поскольку каждая из этих итераций может занять несколько дней (в зависимости от длины очереди в лабораторию), стоит заранее потратить время на отладку: это экономит много времени в долгосрочной перспективе. По мере увеличения мощности облачной лаборатории эта проблема должна стать менее острой. Тем не менее, вряд ли ваш первый протокол будет успешным — здесь слишком много переменных.
""" PCR overlap extension of sfGFP according to NEB protocol.
v5: Use 3/10ths as much primer as the v4 protocol.
v6: more complex touchdown pcr procedure. The Q5 temperature was probably too hot
v7: more time at low temperature to allow gene-specific part to anneal
v8: correct dNTP concentration, real touchdown
"""
p = Protocol()
# ---------------------------------------------------
# Set up experiment
#
experiment_name = "sfgfp_pcroe_v8"
template_length = 740
_options = {'dilute_primers' : False, # if working stock has not been made
'dilute_template': False, # if working stock has not been made
'dilute_dNTP' : False, # if working stock has not been made
'run_gel' : True, # run a gel to see the plasmid size
'run_absorbance' : False, # check absorbance at 260/280/320
'run_sanger' : False} # sanger sequence the new sequence
options = {k for k,v in _options.items() if v is True}
# ---------------------------------------------------
# Inventory and provisioning
# https://developers.transcriptic.com/v1.0/docs/containers
#
# 'sfgfp2': 'ct17yx8h77tkme', # inventory; sfGFP tube #2, micro-1.5, cold_20
# 'sfgfp_puc19_primer1': 'ct17z9542mrcfv', # inventory; micro-2.0, cold_4
# 'sfgfp_puc19_primer2': 'ct17z9542m5ntb', # inventory; micro-2.0, cold_4
# 'sfgfp_idt_1ngul': 'ct184nnd3rbxfr', # inventory; micro-1.5, cold_4, (ERROR: no template)
#
inv = {
'Q5 Polymerase': 'rs16pcce8rdytv', # catalog; Q5 High-Fidelity DNA Polymerase
'Q5 Buffer': 'rs16pcce8rmke3', # catalog; Q5 Reaction Buffer
'dNTP Mixture': 'rs16pcb542c5rd', # catalog; dNTP Mixture (25mM?)
'water': 'rs17gmh5wafm5p', # catalog; Autoclaved MilliQ H2O
'sfgfp_pcroe_v5_puc19_primer1_10uM': 'ct186cj5cqzjmr', # inventory; micro-1.5, cold_4
'sfgfp_pcroe_v5_puc19_primer2_10uM': 'ct186cj5cq536x', # inventory; micro-1.5, cold_4
'sfgfp1': 'ct17yx8h759dk4', # inventory; sfGFP tube #1, micro-1.5, cold_20
}
# Existing inventory
template_tube = p.ref("sfgfp1", id=inv['sfgfp1'], cont_type="micro-1.5", storage="cold_4").well(0)
dilute_primer_tubes = [p.ref('sfgfp_pcroe_v5_puc19_primer1_10uM', id=inv['sfgfp_pcroe_v5_puc19_primer1_10uM'], cont_type="micro-1.5", storage="cold_4").well(0),
p.ref('sfgfp_pcroe_v5_puc19_primer2_10uM', id=inv['sfgfp_pcroe_v5_puc19_primer2_10uM'], cont_type="micro-1.5", storage="cold_4").well(0)]
# New inventory resulting from this experiment
dilute_template_tube = p.ref("sfgfp1_0.25ngul", cont_type="micro-1.5", storage="cold_4").well(0)
dNTP_10uM_tube = p.ref("dNTP_10uM", cont_type="micro-1.5", storage="cold_4").well(0)
sfgfp_pcroe_out_tube = p.ref(expid("amplified"), cont_type="micro-1.5", storage="cold_4").well(0)
# Temporary tubes for use, then discarded
mastermix_tube = p.ref("mastermix", cont_type="micro-1.5", storage="cold_4", discard=True).well(0)
water_tube = p.ref("water", cont_type="micro-1.5", storage="ambient", discard=True).well(0)
pcr_plate = p.ref("pcr_plate", cont_type="96-pcr", storage="cold_4", discard=True)
if 'run_absorbance' in options:
abs_plate = p.ref("abs_plate", cont_type="96-flat", storage="cold_4", discard=True)
# Initialize all existing inventory
all_inventory_wells = [template_tube] + dilute_primer_tubes
for well in all_inventory_wells:
init_inventory_well(well)
print(well.name, well.volume, well.properties)
# -----------------------------------------------------
# Provision water once, for general use
#
p.provision(inv["water"], water_tube, µl(500))
# -----------------------------------------------------
# Dilute primers 1/10 (100uM->10uM) and keep at 4C
#
if 'dilute_primers' in options:
for primer_num in (0,1):
p.transfer(water_tube, dilute_primer_tubes[primer_num], µl(90))
p.transfer(primer_tubes[primer_num], dilute_primer_tubes[primer_num], µl(10), mix_before=True, mix_vol=µl(50))
p.mix(dilute_primer_tubes[primer_num], volume=µl(50), repetitions=10)
# -----------------------------------------------------
# Dilute template 1/10 (10ng/ul->1ng/ul) and keep at 4C
# OR
# Dilute template 1/40 (10ng/ul->0.25ng/ul) and keep at 4C
#
if 'dilute_template' in options:
p.transfer(water_tube, dilute_template_tube, µl(195))
p.mix(dilute_template_tube, volume=µl(100), repetitions=10)
# Dilute dNTP to exactly 10uM
if 'dilute_DNTP' in options:
p.transfer(water_tube, dNTP_10uM_tube, µl(6))
p.provision(inv["dNTP Mixture"], dNTP_10uM_tube, µl(4))
# -----------------------------------------------------
# Q5 PCR protocol
# www.neb.com/protocols/2013/12/13/pcr-using-q5-high-fidelity-dna-polymerase-m0491
#
# 25ul reaction
# -------------
# Q5 reaction buffer 5 µl
# Q5 polymerase 0.25 µl
# 10mM dNTP 0.5 µl -- 1µl = 4x12.5mM
# 10uM primer 1 1.25 µl
# 10uM primer 2 1.25 µl
# 1pg-1ng Template 1 µl -- 0.5 or 1ng/ul concentration
# -------------------------------
# Sum 9.25 µl
#
#
# Mastermix tube will have 96ul of stuff, leaving space for 4x1ul aliquots of template
p.transfer(water_tube, mastermix_tube, µl(64))
p.provision(inv["Q5 Buffer"], mastermix_tube, µl(20))
p.provision(inv['Q5 Polymerase'], mastermix_tube, µl(1))
p.transfer(dNTP_10uM_tube, mastermix_tube, µl(1), mix_before=True, mix_vol=µl(2))
p.transfer(dilute_primer_tubes[0], mastermix_tube, µl(5), mix_before=True, mix_vol=µl(10))
p.transfer(dilute_primer_tubes[1], mastermix_tube, µl(5), mix_before=True, mix_vol=µl(10))
p.mix(mastermix_tube, volume="48:microliter", repetitions=10)
# Transfer mastermix to pcr_plate without template
p.transfer(mastermix_tube, pcr_plate.wells(["A1","B1","C1"]), µl(24))
p.transfer(mastermix_tube, pcr_plate.wells(["A2"]), µl(24)) # acknowledged dead volume problems
p.mix(pcr_plate.wells(["A1","B1","C1","A2"]), volume=µl(12), repetitions=10)
# Finally add template
p.transfer(template_tube, pcr_plate.wells(["A1","B1","C1"]), µl(1))
p.mix(pcr_plate.wells(["A1","B1","C1"]), volume=µl(12.5), repetitions=10)
# ---------------------------------------------------------
# Thermocycle with Q5 and hot start
# 61.1 annealing temperature is recommended by NEB protocol
# p.seal is enforced by transcriptic
#
extension_time = int(max(2, np.ceil(template_length * (11.0/1000))))
assert 0 < extension_time < 60, "extension time should be reasonable for PCR"
cycles = [{"cycles": 1, "steps": [{"temperature": "98:celsius", "duration": "30:second"}]}] +
touchdown(70, 61, [8, 25, extension_time], stepsize=0.5) +
[{"cycles": 16, "steps": [{"temperature": "98:celsius", "duration": "8:second"},
{"temperature": "61.1:celsius", "duration": "25:second"},
{"temperature": "72:celsius", "duration": "{:d}:second".format(extension_time)}]},
{"cycles": 1, "steps": [{"temperature": "72:celsius", "duration": "2:minute"}]}]
p.seal(pcr_plate)
p.thermocycle(pcr_plate, cycles, volume=µl(25))
# --------------------------------------------------------
# Run a gel to hopefully see a 740bp fragment
#
if 'run_gel' in options:
p.unseal(pcr_plate)
p.mix(pcr_plate.wells(["A1","B1","C1","A2"]), volume=µl(12.5), repetitions=10)
p.transfer(pcr_plate.wells(["A1","B1","C1","A2"]), pcr_plate.wells(["D1","E1","F1","D2"]),
[µl(2), µl(4), µl(8), µl(8)])
p.transfer(water_tube, pcr_plate.wells(["D1","E1","F1","D2"]),
[µl(18),µl(16),µl(12),µl(12)], mix_after=True, mix_vol=µl(10))
p.gel_separate(pcr_plate.wells(["D1","E1","F1","D2"]),
µl(20), "agarose(10,2%)", "ladder1", "10:minute", expid("gel"))
#---------------------------------------------------------
# Absorbance dilution series. Take 1ul out of the 25ul pcr plate wells
#
if 'run_absorbance' in options:
p.unseal(pcr_plate)
abs_wells = ["A1","B1","C1","A2","B2","C2","A3","B3","C3"]
p.transfer(water_tube, abs_plate.wells(abs_wells[0:6]), µl(10))
p.transfer(water_tube, abs_plate.wells(abs_wells[6:9]), µl(9))
p.transfer(pcr_plate.wells(["A1","B1","C1"]), abs_plate.wells(["A1","B1","C1"]), µl(1), mix_after=True, mix_vol=µl(5))
p.transfer(abs_plate.wells(["A1","B1","C1"]), abs_plate.wells(["A2","B2","C2"]), µl(1), mix_after=True, mix_vol=µl(5))
p.transfer(abs_plate.wells(["A2","B2","C2"]), abs_plate.wells(["A3","B3","C3"]), µl(1), mix_after=True, mix_vol=µl(5))
for wavelength in [260, 280, 320]:
p.absorbance(abs_plate, abs_plate.wells(abs_wells),
"{}:nanometer".format(wavelength), exp_id("abs_{}".format(wavelength)), flashes=25)
# -----------------------------------------------------------------------------
# Sanger sequencing: https://developers.transcriptic.com/docs/sanger-sequencing
# "Each reaction should have a total volume of 15 µl and we recommend the following composition of DNA and primer:
# PCR product (40 ng), primer (1 µl of a 10 µM stock)"
#
# By comparing to the gel ladder concentration (175ng/lane), it looks like 5ul of PCR product has approximately 30ng of DNA
#
if 'run_sanger' in options:
p.unseal(pcr_plate)
seq_wells = ["G1","G2"]
for primer_num, seq_well in [(0, seq_wells[0]),(1, seq_wells[1])]:
p.transfer(dilute_primer_tubes[primer_num], pcr_plate.wells([seq_well]),
µl(1), mix_before=True, mix_vol=µl(50))
p.transfer(pcr_plate.wells(["A1"]), pcr_plate.wells([seq_well]),
µl(5), mix_before=True, mix_vol=µl(10))
p.transfer(water_tube, pcr_plate.wells([seq_well]), µl(9))
p.mix(pcr_plate.wells(seq_wells), volume=µl(7.5), repetitions=10)
p.sangerseq(pcr_plate, pcr_plate.wells(seq_wells[0]).indices(), expid("seq1"))
p.sangerseq(pcr_plate, pcr_plate.wells(seq_wells[1]).indices(), expid("seq2"))
# -------------------------------------------------------------------------
# Then consolidate to one tube. Leave at least 3ul dead volume in each tube
#
remaining_volumes = [well.volume - dead_volume['96-pcr'] for well in pcr_plate.wells(["A1","B1","C1"])]
print("Consolidated volume", sum(remaining_volumes, µl(0)))
p.consolidate(pcr_plate.wells(["A1","B1","C1"]), sfgfp_pcroe_out_tube, remaining_volumes, allow_carryover=True)
uprint("nProtocol 1. Amplify the insert (oligos previously synthesized)")
jprotocol = json.dumps(p.as_dict(), indent=2)
!echo '{jprotocol}' | transcriptic analyze
open("protocol_{}.json".format(experiment_name),'w').write(jprotocol)WARNING:root:Low volume for well sfGFP 1 /sfGFP 1 : 2.0:microliter
sfGFP 1 /sfGFP 1 2.0:microliter {'dilution': '0.25ng/ul'}
sfgfp_pcroe_v5_puc19_primer1_10uM 75.0:microliter {}
sfgfp_pcroe_v5_puc19_primer2_10uM 75.0:microliter {}
Consolidated volume 52.0:microliter
Protocol 1. Amplify the insert (oligos previously synthesized)
---------------------------------------------------------------
✓ Protocol analyzed
11 instructions
8 containers
Total Cost: $32.18
Workcell Time: $4.32
Reagents & Consumables: $27.86
Результаты: PCR инсерции
Анализ результатов в геле
В геле можно оценить правильный размер продукта после увеличения концентрации (положение полоски в геле) и правильное количество (темнота полоски). В геле есть лестница, соответствующая различным длинам и количествам ДНК, которые можно использовать для сравнения.
На гелевой фотографии ниже полосы D1, E1, F1 содержат соответственно 2 мкл, 4 мкл и 8 мкл амплифицированного продукта. Я могу оценить количество ДНК в каждой полосе по сравнению с ДНК в лестнице (50 нг ДНК на полосу в лестнице). Думаю, что результаты выглядят очень чистыми.
Я попытался использовать для анализа изображения и оценки концентрации GelEval, и вполне успешно, хотя я не уверен, что это намного точнее, чем более наивный метод. Однако небольшие изменения в расположении и размере полос привели к большим изменениям в оценке количества ДНК. Моя лучшая оценка количества ДНК в моём амплифицированном продукте с использованием GelEval составляет 40 нг/мкл.
Если предположить, что мы ограничены количеством праймера в смеси, а не количеством dNTP или фермента, то, поскольку у меня есть 12,5 пмоль каждого праймера, это означает теоретический максимум 6 мкг ДНК 740bp в 25 мкл. Поскольку моя оценка общего количества ДНК с использованием GelEval составляет 40 нг x 25 мкл (1 мкг или 2 пмоля), эти результаты очень разумны и близки к тому, что я должен ожидать в идеальных условиях.

Гель-электрофорез EcoRI-среза pUC19, различные концентрации (D1, E1, F1), плюс контроль (D2)
Диагностика результатов PCR
Недавно Transcriptic начала предоставлять интересные и полезные диагностические данные от своих роботов. На момент написания статьи они недоступны для загрузки, поэтому пока у меня есть только изображение температур во время термоциклирования.
Данные выглядят хорошо, без неожиданных пиков или впадин. В общей сложности 35 циклов PCR, но некоторые из этих циклов проводятся при очень высокой температуре в рамках тачдауна PCR. В моих предыдущих попытках амплифицировать этот сегмент — которых было несколько! — возникали проблемы с гибридизацией праймеров, поэтому здесь PCR довольно много времени работает на высоких температурах, что должно повысить точность.

Термоциклическая диагностика для тачдауна PCR: температуры блока, образца и крышки в течение 35 циклов и 42 минут
Шаг 2. Резка плазмиды
Чтобы вставить нашу ДНК sfGFP в pUC19, сначала нужно разрезать плазмиду. Следуя протоколу NEB, я делаю это с помощью ограничительного фермента EcoRI. В стандартном инвентаре Transcriptic есть реагенты, которые мне нужны: это NEB EcoRI и 10x CutSmart буфер, а также плазмида NEB pUC19.
Для информации, ниже цены из их инвентаря. На самом деле плачу только часть цены, так как Transcriptic берёт оплату за фактически потреблённое количество:
Item ID Amount Concentration Price ------------ ------ ------------- ----------------- ------ CutSmart 10x B7204S 5 ml 10 X $19.00 EcoRI R3101L 50,000 units 20,000 units/ml $225.00 pUC19 N3041L 250 µg 1,000 µg/ml $268.00
Я максимально следовал протоколу NEB:
Перед использованием буфер должен быть полностью разморожен. Разбавьте запас 10X с dH2O до конечной концентрации 1X. Сначала добавьте воду, затем буфер, раствор ДНК и, наконец, фермент. Типичная реакция 50 мкл должна содержать 5 мкл 10x NEBuffer с остальной частью объёма из раствора ДНК, фермента и dH2O.
Одна единица определяется как количество фермента, необходимое для освоения 1 мкг λ ДНК в течение 1 часа при 37°C в общем объёме реакции 50 мкл. В целом, мы рекомендуем 5-10 единиц фермента на мкг ДНК и 10-20 единиц геномной ДНК в 1-часовом процессе.
Для освоения 1 мкг субстрата рекомендуется реакционный объём 50 мкл.
"""Protocol for cutting pUC19 with EcoRI."""
p = Protocol()
experiment_name = "puc19_ecori_v3"
options = {}
inv = {
'water': "rs17gmh5wafm5p", # catalog; Autoclaved MilliQ H2O; ambient
"pUC19": "rs17tcqmncjfsh", # catalog; pUC19; cold_20
"EcoRI": "rs17ta8xftpdk6", # catalog; EcoRI-HF; cold_20
"CutSmart": "rs17ta93g3y85t", # catalog; CutSmart Buffer 10x; cold_20
"ecori_p10x": "ct187v4ea85k2h", # inventory; EcoRI diluted 10x
}
# Tubes and plates I use then discard
re_tube = p.ref("re_tube", cont_type="micro-1.5", storage="cold_4", discard=True).well(0)
water_tube = p.ref("water_tube", cont_type="micro-1.5", storage="cold_4", discard=True).well(0)
pcr_plate = p.ref("pcr_plate", cont_type="96-pcr", storage="cold_4", discard=True)
# The result of the experiment, a pUC19 cut by EcoRI, goes in this tube for storage
puc19_cut_tube = p.ref(expid("puc19_cut"), cont_type="micro-1.5", storage="cold_20").well(0)
# -------------------------------------------------------------
# Provisioning and diluting.
# Diluted EcoRI can be used more than once
#
p.provision(inv["water"], water_tube, µl(500))
if 'dilute_ecori' in options:
ecori_p10x_tube = p.ref("ecori_p10x", cont_type="micro-1.5", storage="cold_20").well(0)
p.transfer(water_tube, ecori_p10x_tube, µl(45))
p.provision(inv["EcoRI"], ecori_p10x_tube, µl(5))
else:
# All "inventory" (stuff I own at transcriptic) must be initialized
ecori_p10x_tube = p.ref("ecori_p10x", id=inv["ecori_p10x"], cont_type="micro-1.5", storage="cold_20").well(0)
init_inventory_well(ecori_p10x_tube)
# -------------------------------------------------------------
# Restriction enzyme cutting pUC19
#
# 50ul total reaction volume for cutting 1ug of DNA:
# 5ul CutSmart 10x
# 1ul pUC19 (1ug of DNA)
# 1ul EcoRI (or 10ul diluted EcoRI, 20 units, >10 units per ug DNA)
#
p.transfer(water_tube, re_tube, µl(117))
p.provision(inv["CutSmart"], re_tube, µl(15))
p.provision(inv["pUC19"], re_tube, µl(3))
p.mix(re_tube, volume=µl(60), repetitions=10)
assert re_tube.volume == µl(120) + dead_volume["micro-1.5"]
print("Volumes: re_tube:{} water_tube:{} EcoRI:{}".format(re_tube.volume, water_tube.volume, ecori_p10x_tube.volume))
p.distribute(re_tube, pcr_plate.wells(["A1","B1","A2"]), µl(40))
p.distribute(water_tube, pcr_plate.wells(["A2"]), µl(10))
p.distribute(ecori_p10x_tube, pcr_plate.wells(["A1","B1"]), µl(10))
assert all(well.volume == µl(50) for well in pcr_plate.wells(["A1","B1","A2"]))
p.mix(pcr_plate.wells(["A1","B1","A2"]), volume=µl(25), repetitions=10)
# Incubation to induce cut, then heat inactivation of EcoRI
p.seal(pcr_plate)
p.incubate(pcr_plate, "warm_37", "60:minute", shaking=False)
p.thermocycle(pcr_plate, [{"cycles": 1, "steps": [{"temperature": "65:celsius", "duration": "21:minute"}]}], volume=µl(50))
# --------------------------------------------------------------
# Gel electrophoresis, to ensure the cutting worked
#
p.unseal(pcr_plate)
p.mix(pcr_plate.wells(["A1","B1","A2"]), volume=µl(25), repetitions=5)
p.transfer(pcr_plate.wells(["A1","B1","A2"]), pcr_plate.wells(["D1","E1","D2"]), µl(8))
p.transfer(water_tube, pcr_plate.wells(["D1","E1","D2"]), µl(15), mix_after=True, mix_vol=µl(10))
assert all(well.volume == µl(20) + dead_volume["96-pcr"] for well in pcr_plate.wells(["D1","E1","D2"]))
p.gel_separate(pcr_plate.wells(["D1","E1","D2"]), µl(20), "agarose(10,2%)", "ladder2", "15:minute", expid("gel"))
# ----------------------------------------------------------------------------
# Then consolidate all cut plasmid to one tube (puc19_cut_tube).
#
remaining_volumes = [well.volume - dead_volume['96-pcr'] for well in pcr_plate.wells(["A1","B1"])]
print("Consolidated volume: {}".format(sum(remaining_volumes, µl(0))))
p.consolidate(pcr_plate.wells(["A1","B1"]), puc19_cut_tube, remaining_volumes, allow_carryover=True)
assert all(tube.volume >= dead_volume['micro-1.5'] for tube in [water_tube, re_tube, puc19_cut_tube, ecori_p10x_tube])
# ---------------------------------------------------------------
# Test protocol
#
jprotocol = json.dumps(p.as_dict(), indent=2)
!echo '{jprotocol}' | transcriptic analyze
#print("Protocol {}nn{}".format(experiment_name, jprotocol))
open("protocol_{}.json".format(experiment_name),'w').write(jprotocol)Volumes: re_tube:135.0:microliter water_tube:383.0:microliter EcoRI:30.0:microliter Consolidated volume: 78.0:microliter ✓ Protocol analyzed 12 instructions 5 containers Total Cost: $30.72 Workcell Time: $3.38 Reagents & Consumables: $27.34
Результаты: резка плазмиды
Я провёл этот эксперимент дважды в немного разных условиях и с гелями разного размера, но результаты почти идентичны. Оба геля мне нравятся.
Первоначально я не выделял достаточно места для «мёртвого» объёма (в пробирках 1,5 мл мёртвый объём 15 мкл!). Думаю, это объясняет разницу между D1 и E1 (эти две полосы должны быть идентичными). Проблему мёртвого объёма легко решить, создав надлежащий рабочий запаса разбавленной EcoRI в начале протокола.
Несмотря на эту ошибку, в обоих гелях полосы D1 и E1 сильные полосы в правильном положении 2,6kb. На полосе D2 неразрезанная плазмида: как и положено, она не видна в одном геле и едва видна в другом.
Две гелевые фотографии выглядят довольно по-разному. Частично это объясняется тем, что данный шаг Transcriptic пока не автоматизировала.

Два геля, показывающие разрезанную pUC19 (2,6kb) на полосах D1 и E1, а неразрезанную pUC19 на D2
Шаг 3. Сборка методом Гибсона
Самый простой способ проверить, работает ли моя сборка методом Гибсона — собрать инсерцию и плазмиду, затем использовать стандартные праймеры M13 (которые фланкируют инсерцию) для амплификации части плазмиды и вставленной ДНК и запустить qPCR и гель, чтобы убедиться, что амплификация сработала. Вы также можете запустить реакцию секвенирования, чтобы подтвердить, что всё инсерцировано как ожидалось, но я решил оставить это на потом.
Если сборка Гибсона не сработает, то амплификация M13 не сработает, потому что плазмида была разрезана между двумя последовательностями M13.
"""Debugging transformation protocol: Gibson assembly followed by qPCR and a gel
v2: include v3 Gibson assembly"""
p = Protocol()
options = {}
experiment_name = "debug_sfgfp_puc19_gibson_seq_v2"
inv = {
"water" : "rs17gmh5wafm5p", # catalog; Autoclaved MilliQ H2O; ambient
"M13_F" : "rs17tcpqwqcaxe", # catalog; M13 Forward (-41); cold_20 (1ul = 100pmol)
"M13_R" : "rs17tcph6e2qzh", # catalog; M13 Reverse (-48); cold_20 (1ul = 100pmol)
"SensiFAST_SYBR_No-ROX" : "rs17knkh7526ha", # catalog; SensiFAST SYBR for qPCR
"sfgfp_puc19_gibson_v1_clone" : "ct187rzdq9kd7q", # inventory; assembled sfGFP; cold_4
"sfgfp_puc19_gibson_v3_clone" : "ct188ejywa8jcv", # inventory; assembled sfGFP; cold_4
}
# ---------------------------------------------------------------
# First get my sfGFP pUC19 clones, assembled with Gibson assembly
#
clone_plate1 = p.ref("sfgfp_puc19_gibson_v1_clone", id=inv["sfgfp_puc19_gibson_v1_clone"],
cont_type="96-pcr", storage="cold_4", discard=False)
clone_plate2 = p.ref("sfgfp_puc19_gibson_v3_clone", id=inv["sfgfp_puc19_gibson_v3_clone"],
cont_type="96-pcr", storage="cold_4", discard=False)
water_tube = p.ref("water", cont_type="micro-1.5", storage="cold_4", discard=True).well(0)
master_tube = p.ref("master", cont_type="micro-1.5", storage="cold_4", discard=True).well(0)
primer_tube = p.ref("primer", cont_type="micro-1.5", storage="cold_4", discard=True).well(0)
pcr_plate = p.ref(expid("pcr_plate"), cont_type="96-pcr", storage="cold_4", discard=False)
init_inventory_well(clone_plate1.well("A1"))
init_inventory_well(clone_plate2.well("A1"))
seq_wells = ["B2","B4","B6", # clone_plate1
"D2","D4","D6", # clone_plate2
"F2","F4"] # control
# clone_plate2 was diluted 4X (20ul->80ul), according to NEB instructions
assert clone_plate1.well("A1").volume == µl(18), clone_plate1.well("A1").volume
assert clone_plate2.well("A1").volume == µl(78), clone_plate2.well("A1").volume
# --------------------------------------------------------------
# Provisioning
#
p.provision(inv["water"], water_tube, µl(500))
# primers, diluted 2X, discarded at the end
p.provision(inv["M13_F"], primer_tube, µl(13))
p.provision(inv["M13_R"], primer_tube, µl(13))
p.transfer(water_tube, primer_tube, µl(26), mix_after=True, mix_vol=µl(20), repetitions=10)
# -------------------------------------------------------------------
# PCR Master mix -- 10ul SYBR mix, plus 1ul each undiluted primer DNA (100pmol)
# Also add 15ul of dead volume
#
p.provision(inv['SensiFAST_SYBR_No-ROX'], master_tube, µl(11+len(seq_wells)*10))
p.transfer(primer_tube, master_tube, µl(4+len(seq_wells)*4))
p.mix(master_tube, volume=µl(63), repetitions=10)
assert master_tube.volume == µl(127) # 15ul dead volume
p.distribute(master_tube, pcr_plate.wells(seq_wells), µl(14), allow_carryover=True)
p.distribute(water_tube, pcr_plate.wells(seq_wells),
[µl(ul) for ul in [5,4,2, 4,2,0, 6,6]],
allow_carryover=True)
# Template -- starting with some small, unknown amount of DNA produced by Gibson
p.transfer(clone_plate1.well("A1"), pcr_plate.wells(seq_wells[0:3]), [µl(1),µl(2),µl(4)], one_tip=True)
p.transfer(clone_plate2.well("A1"), pcr_plate.wells(seq_wells[3:6]), [µl(2),µl(4),µl(6)], one_tip=True)
assert all(pcr_plate.well(w).volume == µl(20) for w in seq_wells)
assert clone_plate1.well("A1").volume == µl(11)
assert clone_plate2.well("A1").volume == µl(66)
# --------------------------------------------------------------
# qPCR
# standard melting curve parameters
#
p.seal(pcr_plate)
p.thermocycle(pcr_plate, [{"cycles": 1, "steps": [{"temperature": "95:celsius","duration": "2:minute"}]},
{"cycles": 40, "steps": [{"temperature": "95:celsius","duration": "5:second"},
{"temperature": "60:celsius","duration": "20:second"},
{"temperature": "72:celsius","duration": "15:second", "read": True}]}],
volume=µl(20), # volume is optional
dataref=expid("qpcr"),
dyes={"SYBR": seq_wells}, # dye must be specified (tells transcriptic what aborbance to use?)
melting_start="65:celsius", melting_end="95:celsius", melting_increment="0.5:celsius", melting_rate="5:second")
# --------------------------------------------------------------
# Gel -- 20ul required
# Dilute such that I have 11ul for sequencing
#
p.unseal(pcr_plate)
p.distribute(water_tube, pcr_plate.wells(seq_wells), µl(11))
p.gel_separate(pcr_plate.wells(seq_wells), µl(20), "agarose(8,0.8%)", "ladder1", "10:minute", expid("gel"))
# This appears to be a bug in Transcriptic. The actual volume should be 11ul
# but it is not updating after running a gel with 20ul.
# Primer tube should be equal to dead volume, or it's a waste
assert all(pcr_plate.well(w).volume==µl(31) for w in seq_wells)
assert primer_tube.volume == µl(16) == dead_volume['micro-1.5'] + µl(1)
assert water_tube.volume > µl(25)
# ---------------------------------------------------------------
# Test and run protocol
#
jprotocol = json.dumps(p.as_dict(), indent=2)
!echo '{jprotocol}' | transcriptic analyze
open("protocol_{}.json".format(experiment_name),'w').write(jprotocol)WARNING:root:Low volume for well sfgfp_puc19_gibson_v1_clone/sfgfp_puc19_gibson_v1_clone : 11.0:microliter
✓ Protocol analyzed 11 instructions 6 containers Total Cost: $32.09 Workcell Time: $6.98 Reagents & Consumables: $25.11
Результаты: qPCR для сборки Гибсона
Я могу получить доступ к данным qPCR в формате JSON через Transcriptic API. Эта функция не очень хорошо документирована, но может быть чрезвычайно полезной. API даже дают вам доступ к некоторым диагностическим данным от роботов, что может помочь в отладке.
Во-первых, запрашиваем данные о запуске:
project_id, run_id = "p16x6gna8f5e9", "r18mj3cz3fku7"
api_url = "https://secure.transcriptic.com/hgbrian/{}/runs/{}/data.json".format(project_id, run_id)
data_response = requests.get(api_url, headers=tsc_headers)
data = data_response.json()Затем указываем этот id, чтобы получить данные «постобработки» qPCR:
qpcr_id = data['debug_sfgfp_puc19_gibson_seq_v1_qpcr']['id']
pp_api_url = "https://secure.transcriptic.com/data/{}.json?key=postprocessed_data".format(qpcr_id)
data_response = requests.get(pp_api_url, headers=tsc_headers)
pp_data = data_response.json()Вот значения Ct (порог цикла) для каждой пробирки. Ct — это просто точка, в которой флуоресценция превышает определённое значение. Она приблизительно говорит, сколько в данный момент там есть ДНК (и, следовательно, приблизительно, с чего мы начали).
# Simple util to convert wellnum to wellname
n_w = {str(wellnum):'ABCDEFGH'[wellnum//12]+str(1+wellnum%12) for wellnum in range(96)}
w_n = {v: k for k, v in n_w.items()}
ct_vals = {n_w[k]:v for k,v in pp_data["amp0"]["SYBR"]["cts"].items()}
ct_df = pd.DataFrame(ct_vals, index=["Ct"]).T
ct_df["well"] = ct_df.index
f, ax = plt.subplots(figsize=(16,6))
_ = sns.barplot(y="well", x="Ct", data=ct_df)
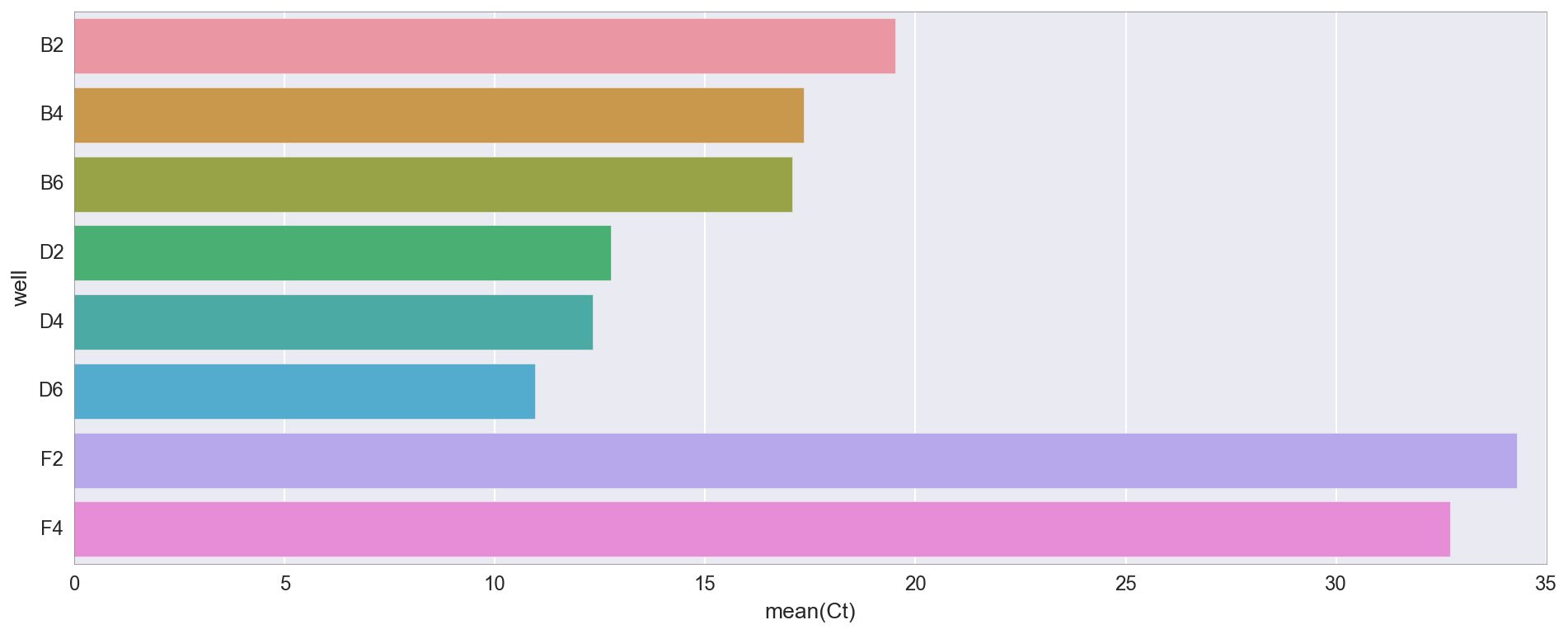
Как видим, амплификация раньше всего происходит в пробирках D2/4/6 (где ДНК из моей сборки Гибсона «v3»), затем B2/4/6 (сборка Гибсона «v1»). Различия между v1 и v3 в основном заключаются в том, что ДНК v3 разбавлена 4X в соответствии с протоколом NEB, но оба варианта должны работать. Существует некоторая амплификация после цикла 30 в контрольных пробирках (F2, F4), не имеющих шаблонной ДНК, но это не редкость, так как они включают в себя много праймерной ДНК.
Я также могу построить кривую амплификации qPCR, чтобы посмотреть динамику амплификации.
f, ax = plt.subplots(figsize=(16,6))
ax.set_color_cycle(['#fb6a4a', '#de2d26', '#a50f15', '#74c476', '#31a354', '#006d2c', '#08519c', '#6baed6'])
amp0 = pp_data['amp0']['SYBR']['baseline_subtracted']
_ = [plt.plot(amp0[w_n[well]], label=well) for well in ['B2', 'B4', 'B6', 'D2', 'D4', 'D6', 'F2', 'F4']]
_ = ax.set_ylim(0,)
_ = plt.title("qPCR (reds=Gibson v1, greens=Gibson v3, blues=control)")
_ = plt.legend(bbox_to_anchor=(1, .75), bbox_transform=plt.gcf().transFigure)

В целом, результаты qPCR выглядят великолепно, с хорошей амплификацией обеих версий моей сборки Гибсона и без реальной амплификации в контрольной группе. Поскольку сборка v3 показала немного лучший результат, чем v1, теперь будем использовать её.
Результаты: сборка Гибсона в геле
Гель также очень чистый, показывает сильные полосы чуть ниже 1kb в полосах B2, B4, B6, D2, D4, D6: именно такой размер мы ожидаем (инсерция составляет около 740bp, а праймеры M13 — около 40bp вверх и вниз). Вторая полоса соответствует праймерам. Можно быть уверенным в этом, так как полосы F2 и F4 содержат только праймерную ДНК.
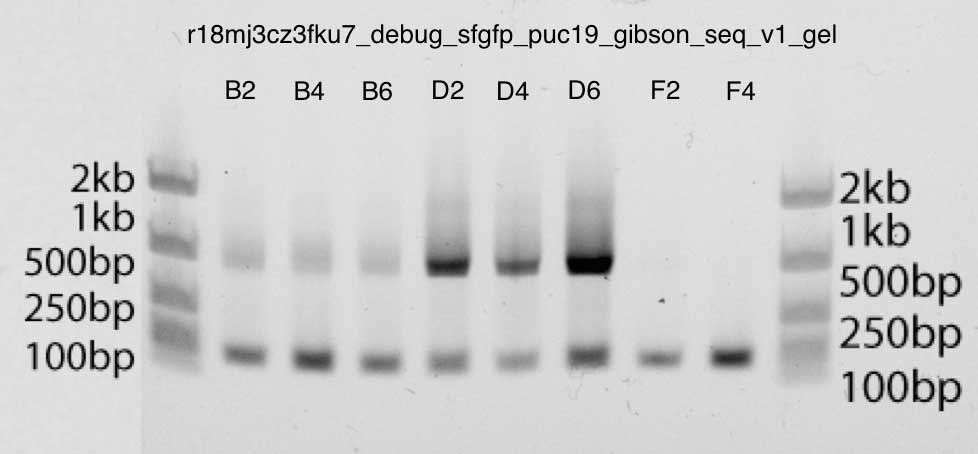
Электрофорез в полиакриламидном геле: с сборка Гибсона v3 показывает более сильные полосы (D2, D4, D6), в соответствии с приведенными выше данными qPCR
Шаг 4. Трансформация
Трансформация — это процесс изменения организма путём добавления ДНК. В этом эксперименте мы трансформируем E. coli с помощью sfGFP-экспрессирующей плазмиды pUC19.
Используем простой в работе штамм Zymo DH5α Mix&Go и рекомендуемый протокол. Этот штамм — часть стандартного инвентаря Transcriptic. В общем, трансформации могут быть сложными, поскольку компетентные ячейки довольно хрупки, поэтому чем проще и надёжнее протокол, тем лучше. В обычных лабораториях молекулярной биологии эти компетентные клетки, вероятно, были бы слишком дороги для общего использования.
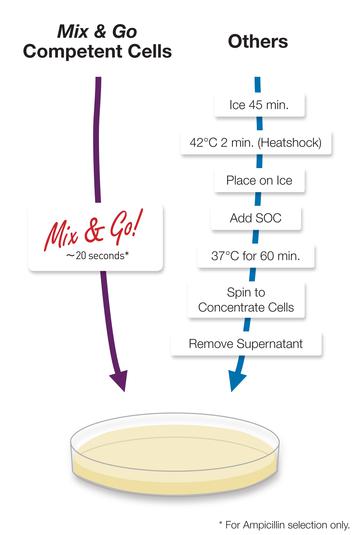
Клетки Zymo Mix & Go с простым протоколом
Проблема с роботами
Этот протокол — хороший пример, насколько трудна адаптация человеческих протоколов для использования роботами и как она может неожиданно потерпеть неудачу. Протоколы иногда на удивление расплывчаты («раскачать пробирку из стороны в сторону»), опираясь на общий контекст молекулярных биологов, или они могут внезапно запросить продвинутую обработку изображения («убедитесь, что осадок размешан»). Люди не возражают против таких задач, но роботам нужны более чёткие инструкции.
В этой трансформации проявились интересные проблемы с таймингом. Протокол трансформации советует, чтобы клетки не оставались при комнатной температуре более нескольких секунд а планшет с пробирками предварительно нагреть до 37°C. Теоретически, вы хотели бы начать предварительное нагревание, чтобы оно закончилось одновременно с трансформацией, но неясно, как роботы Transcriptic справятся с этой ситуацией — насколько мне известно, нет способа точно синхронизировать шаги протокола. Отсутствие точного контроля над временем, похоже, будет общей проблемой с роботизированными протоколами из-за сравнительной негибкости роботизированной руки, конфликтов планирования и т. д. Нам придётся соответствующим образом скорректировать протоколы.
Обычно есть разумные решения: иногда вам просто нужно использовать разные реагенты (например, более выносливые клетки, такие как Mix&Go выше); иногда вы просто закладываете действия с запасом (например, встряхнуть десять раз вместо трёх); иногда нужно придумать для роботов специальные трюки (например, использовать машину PCR для теплового удара).
Конечно, большим преимуществом является то, что, как только протокол сработал однажды, можно в целом полагаться на него снова и снова. Вы даже можете количественно оценить, насколько надёжен протокол, и со временем улучшить его!
Тестовая трансформация
Прежде чем начать трансформацию с полностью собранной плазмидой, я провожу простой эксперимент, чтобы убедиться, что трансформация с использованием обычного pUC19 (т. е. без сборки Гибсона и без инсерции ДНК sfGFP) работает. pUC19 содержит ген устойчивости к ампициллину, поэтому успешная трансформация должна позволить бактериям расти на пластинах, содержащих этот антибиотик.
Я переношу бактерии прямо на пластины («6-flat» в терминологии Transcriptic), где либо есть ампициллин, либо нет. Я ожидаю, что трансформированные бактерии содержат ген устойчивости к ампициллину и, следовательно, будут расти. Нетрансформированные бактерии не должны расти.
"""Simple transformation protocol: transformation with unaltered pUC19"""
p = Protocol()
experiment_name = "debug_sfgfp_puc19_gibson_v1"
inv = {
"water" : "rs17gmh5wafm5p", # catalog; Autoclaved MilliQ H2O; ambient
"DH5a" : "rs16pbj944fnny", # catalog; Zymo DH5α; cold_80
"LB Miller" : "rs17bafcbmyrmh", # catalog; LB Broth Miller; cold_4
"Amp 100mgml" : "rs17msfk8ujkca", # catalog; Ampicillin 100mg/ml; cold_20
"pUC19" : "rs17tcqmncjfsh", # catalog; pUC19; cold_20
}
# Catalog
transform_plate = p.ref("transform_plate", cont_type="96-pcr", storage="ambient", discard=True)
transform_tube = transform_plate.well(0)
# ------------------------------------------------------------------------------------
# Plating transformed bacteria according to Tali's protocol (requires different code!)
# http://learn.transcriptic.com/blog/2015/9/9/provisioning-commercial-reagents
# Add 1-5ul plasmid and pre-warm culture plates to 37C before starting.
#
#
# Extra inventory for plating
#
inv["lb-broth-100ug-ml-amp_6-flat"] = "ki17sbb845ssx9" # (kit, not normal ref) from blogpost
inv["noAB-amp_6-flat"] = "ki17reefwqq3sq" # kit id
inv["LB Miller"] = "rs17bafcbmyrmh"
#
# Ampicillin and no ampicillin plates
#
amp_6_flat = Container(None, p.container_type('6-flat'))
p.refs["amp_6_flat"] = Ref('amp_6_flat',
{"reserve": inv['lb-broth-100ug-ml-amp_6-flat'], "store": {"where": 'cold_4'}}, amp_6_flat)
noAB_6_flat = Container(None, p.container_type('6-flat'))
p.refs["noAB_6_flat"] = Ref('noAB_6_flat',
{"reserve": inv['noAB-amp_6-flat'], "store": {"where": 'cold_4'}}, noAB_6_flat)
#
# Provision competent bacteria
#
p.provision(inv["DH5a"], transform_tube, µl(50))
p.provision(inv["pUC19"], transform_tube, µl(2))
#
# Heatshock the bacteria to transform using a PCR machine
#
p.seal(transform_plate)
p.thermocycle(transform_plate,
[{"cycles": 1, "steps": [{"temperature": "4:celsius", "duration": "5:minute"}]},
{"cycles": 1, "steps": [{"temperature": "37:celsius", "duration": "30:minute"}]}],
volume=µl(50))
p.unseal(transform_plate)
#
# Then dilute bacteria and spread onto 6-flat plates
# Put more on ampicillin plates for more opportunities to get a colony
#
p.provision(inv["LB Miller"], transform_tube, µl(355))
p.mix(transform_tube, µl(150), repetitions=5)
for i in range(6):
p.spread(transform_tube, amp_6_flat.well(i), µl(55))
p.spread(transform_tube, noAB_6_flat.well(i), µl(10))
assert transform_tube.volume >= µl(15), transform_tube.volume
#
# Incubate and image 6-flat plates over 18 hours
#
for flat_name, flat in [("amp_6_flat", amp_6_flat), ("noAB_6_flat", noAB_6_flat)]:
for timepoint in [6,12,18]:
p.cover(flat)
p.incubate(flat, "warm_37", "6:hour")
p.uncover(flat)
p.image_plate(flat, mode="top", dataref=expid("{}_t{}".format(flat_name, timepoint)))
# ---------------------------------------------------------------
# Analyze protocol
#
jprotocol = json.dumps(p.as_dict(), indent=2)
!echo '{jprotocol}' | transcriptic analyze
#print("Protocol {}nn{}".format(experiment_name, protocol))
open("protocol_{}.json".format(experiment_name),'w').write(jprotocol)✓ Protocol analyzed 43 instructions 3 containers $45.43
Результаты: тестовая трансформация
На следующих фотографиях мы видим, что без антибиотика (пластины слева) на всех шести пластинах наблюдается рост, хотя сильно в разной степени, что вызывает беспокойство. Похоже, роботы Transcriptic не особо справляются с равномерным распределением, что требует некоторой ловкости.
В присутствии антибиотика (пластины справа) тоже есть рост, хотя он снова непоследователен. Первые две пластины с антибиотиками выглядят странно, с большим ростом, что, вероятно, является результатом добавления 55 мкл на эти пластины по сравнению с 10 мкл на пластинах без антибиотиков. На третьей пластине несколько колоний и по существу такое я ожидал увидеть на всех пластинах. На последних трёх пластинах должен быть некоторый рост, но его не происходит. Единственное моё объяснение этим странным результатам в том, что я недостаточно смешал клетки и среду, поэтому почти все клетки попали в первые две пластины.
(Нужно было ещё сделать положительный контроль на ампициллин с неизменённой бактерией, но я уже сделал это в предыдущем эксперименте, так что я знаю, что стоковый ампициллин должен убить этот штамм E. coli. Рост гораздо слабее на пластинах с ампициллином, хотя там гораздо больше бактерий, как и предполагалось).
В целом, трансформация сработала достаточно хорошо, чтобы продолжить, хотя есть некоторые изъяны.

Пластины клеток, трансформированных pUC19, через 18 часов: без антибиотика (слева) и с антибиотиком (справа)
Трансформация продукта после сборки
Поскольку сборка Гибсона и простая трансформация pUC19, похоже, работают, теперь можно попробовать трансформацию с полностью собранной плазмидой, экспрессирующей sfGFP.
Помимо собранной инсерции, я также добавлю на пластины немного IPTG и X-gal, чтобы увидеть успешное преобразование на сине-белом экране. Эта дополнительная информация полезна, так как если пройдёт трансформация с обычным pUC19, который не содержит sfGFP, он всё равно даст устойчивость к антибиотикам.
Поглощение и флуоресценция
Согласно этой таблице, sfGFP лучше всего светится на длинах волны возбуждения 485 нм / 510 нм. Я обнаружил, что в Transcriptic лучше работает 485/535. Предполагаю, потому что 485 и 510 слишком похожи. Я измеряю рост бактерий на 600 нм (OD600).
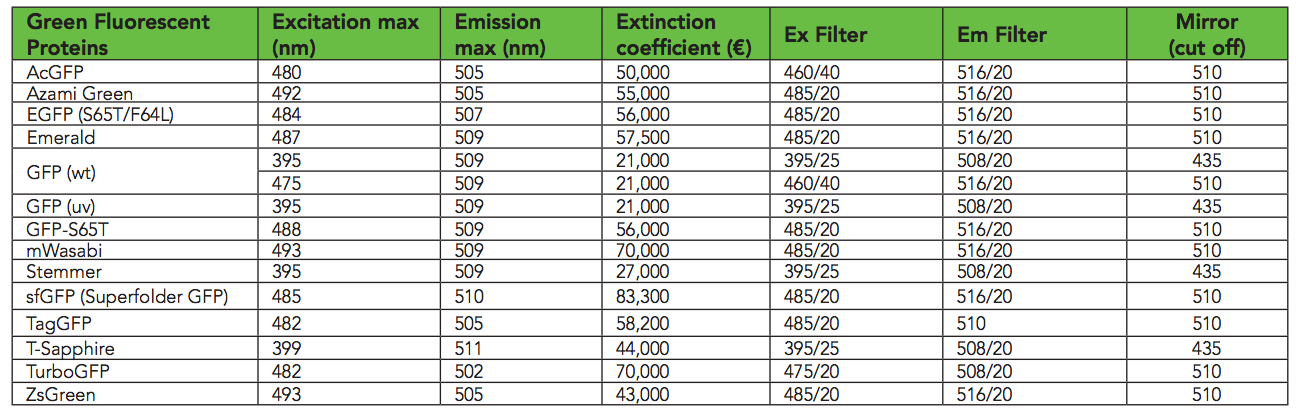
Разнообразие GFP (biotek)
IPTG и X-gal
Мой IPTG находится в концентрации 1M и должен разбавляться 1:1000. В свою очередь, X-gal в концентрации 20 мг/мл тоже следует разбавлять 1:1000 (20 мг/мкл). Следовательно, на 2000µl LB я добавляю по 2 мкл каждого.
Согласно одному протоколу нужно сначала взять 40 мкл X-gal в концентрации 20 мг/мл и 40 мкл IPTG концентрации 0,1 mM (или 4 мкл IPTG на 1M), и выдержать 30 минут. Эта процедура у меня не сработала, поэтому я просто смешал IPTG, X-gal и соответствующие клетки и использовал напрямую эту смесь.
"""Full Gibson assembly and transformation protocol for sfGFP and pUC19
v1: Spread IPTG and X-gal onto plates, then spread cells
v2: Mix IPTG, X-gal and cells; spread the mixture
v3: exclude X-gal so I can do colony picking better
v4: repeat v3 to try other excitation/emission wavelengths"""
p = Protocol()
options = {
"gibson" : False, # do a new gibson assembly
"sanger" : False, # sanger sequence product
"control_pUC19" : True, # unassembled pUC19
"XGal" : False # excluding X-gal should make the colony picking easier
}
for k, v in list(options.items()):
if v is False: del options[k]
experiment_name = "sfgfp_puc19_gibson_plates_v4"
# -----------------------------------------------------------------------
# Inventory
#
inv = {
# catalog
"water" : "rs17gmh5wafm5p", # catalog; Autoclaved MilliQ H2O; ambient
"DH5a" : "rs16pbj944fnny", # catalog; Zymo DH5α; cold_80
"Gibson Mix" : "rs16pfatkggmk5", # catalog; Gibson Mix (2X); cold_20
"LB Miller" : "rs17bafcbmyrmh", # catalog; LB Broth Miller; cold_4
"Amp 100mgml" : "rs17msfk8ujkca", # catalog; Ampicillin 100mg/ml; cold_20
"pUC19" : "rs17tcqmncjfsh", # catalog; pUC19; cold_20
# my inventory
"puc19_cut_v2": "ct187v4ea7vvca", # inventory; pUC19 cut with EcoRI; cold_20
"IPTG" : "ct18a2r5wn6tqz", # inventory; IPTG at 1M (conc semi-documented); cold_20
"XGal" : "ct18a2r5wp5hcv", # inventory; XGal at 0.1M (conc not documented); cold_20
"sfgfp_pcroe_v8_amplified" : "ct1874zqh22pab", # inventory; sfGFP amplified to 40ng/ul; cold_4
"sfgfp_puc19_gibson_v3_clone" : "ct188ejywa8jcv", # inventory; assembled sfGFP; cold_4
# kits (must be used differently)
"lb-broth-100ug-ml-amp_6-flat" : "ki17sbb845ssx9", # catalog; ampicillin plates
"noAB-amp_6-flat" : "ki17reefwqq3sq" # catalog; no antibiotic plates
}
#
# Catalog (all to be discarded afterward)
#
water_tube = p.ref("water", cont_type="micro-1.5", storage="ambient", discard=True).well(0)
transform_plate = p.ref("trn_plate", cont_type="96-pcr", storage="ambient", discard=True)
transform_tube = transform_plate.well(39) # experiment
transform_tube_L = p.ref("trn_tubeL", cont_type="micro-1.5", storage="ambient", discard=True).well(0)
transctrl_tube = transform_plate.well(56) # control
transctrl_tube_L = p.ref("trc_tubeL", cont_type="micro-1.5", storage="ambient", discard=True).well(0)
#
# Plating according to Tali's protocol
# http://learn.transcriptic.com/blog/2015/9/9/provisioning-commercial-reagents
#
amp_6_flat = Container(None, p.container_type('6-flat'))
p.refs[expid("amp_6_flat")] = Ref(expid("amp_6_flat"),
{"reserve": inv['lb-broth-100ug-ml-amp_6-flat'], "store": {"where": 'cold_4'}}, amp_6_flat)
noAB_6_flat = Container(None, p.container_type('6-flat'))
p.refs[expid("noAB_6_flat")] = Ref(expid("noAB_6_flat"),
{"reserve": inv['noAB-amp_6-flat'], "store": {"where": 'cold_4'}}, noAB_6_flat)
#
# My inventory: EcoRI-cut pUC19, oePCR'd sfGFP, Gibson-assembled pUC19, IPTG and X-Gal
#
if "gibson" in options:
puc19_cut_tube = p.ref("puc19_ecori_v2_puc19_cut", id=inv["puc19_cut_v2"],
cont_type="micro-1.5", storage="cold_20").well(0)
sfgfp_pcroe_amp_tube = p.ref("sfgfp_pcroe_v8_amplified", id=inv["sfgfp_pcroe_v8_amplified"],
cont_type="micro-1.5", storage="cold_4").well(0)
clone_plate = p.ref(expid("clone"), cont_type="96-pcr", storage="cold_4", discard=False)
else:
clone_plate = p.ref("sfgfp_puc19_gibson_v3_clone", id=inv["sfgfp_puc19_gibson_v3_clone"],
cont_type="96-pcr", storage="cold_4", discard=False)
IPTG_tube = p.ref("IPTG", id=inv["IPTG"], cont_type="micro-1.5", storage="cold_20").well(0)
if "XGal" in options: XGal_tube = p.ref("XGal", id=inv["XGal"], cont_type="micro-1.5", storage="cold_20").well(0)
#
# Initialize inventory
#
if "gibson" in options:
all_inventory_wells = [puc19_cut_tube, sfgfp_pcroe_amp_tube, IPTG_tube]
assert puc19_cut_tube.volume == µl(66), puc19_cut_tube.volume
assert sfgfp_pcroe_amp_tube.volume == µl(36), sfgfp_pcroe_amp_tube.volume
else:
all_inventory_wells = [IPTG_tube, clone_plate.well(0)]
if "XGal" in options: all_inventory_wells.append(XGal_tube)
for well in all_inventory_wells:
init_inventory_well(well)
print("Inventory: {} {} {}".format(well.name, well.volume, well.properties))
#
# Provisioning. Water is used all over the protocol. Provision an excess since it's cheap
#
p.provision(inv["water"], water_tube, µl(500))
# -----------------------------------------------------------------------------
# Cloning/assembly (see NEBuilder protocol above)
#
# "Optimized efficiency is 50–100 ng of vectors with 2 fold excess of inserts."
# pUC19 is 20ng/ul (78ul total).
# sfGFP is ~40ng/ul (48ul total)
# Therefore 4ul of each gives 80ng and 160ng of vector and insert respectively
#
def do_gibson_assembly():
#
# Combine all the Gibson reagents in one tube and thermocycle
#
p.provision(inv["Gibson Mix"], clone_plate.well(0), µl(10))
p.transfer(water_tube, clone_plate.well(0), µl(2))
p.transfer(puc19_cut_tube, clone_plate.well(0), µl(4))
p.transfer(sfgfp_pcroe_amp_tube, clone_plate.well(0), µl(4),
mix_after=True, mix_vol=µl(10), repetitions=10)
p.seal(clone_plate)
p.thermocycle(clone_plate,
[{"cycles": 1, "steps": [{"temperature": "50:celsius", "duration": "16:minute"}]}],
volume=µl(50))
#
# Dilute assembled plasmid 4X according to the NEB Gibson assembly protocol (20ul->80ul)
#
p.unseal(clone_plate)
p.transfer(water_tube, clone_plate.well(0), µl(60), mix_after=True, mix_vol=µl(40), repetitions=5)
return
# --------------------------------------------------------------------------------------------------
# Transformation
# "Transform NEB 5-alpha Competent E. coli cells with 2 μl of the
# assembled product, following the appropriate transformation protocol."
#
# Mix & Go http://www.zymoresearch.com/downloads/dl/file/id/173/t3015i.pdf
# "[After mixing] Immediately place on ice and incubate for 2-5 minutes"
# "The highest transformation efficiencies can be obtained by incubating Mix & Go cells with DNA on
# ice for 2-5 minutes (60 minutes maximum) prior to plating."
# "It is recommended that culture plates be pre-warmed to >20°C (preferably 37°C) prior to plating."
# "Avoid exposing the cells to room temperature for more than a few seconds at a time."
#
# "If competent cells are purchased from other manufacture, dilute assembled products 4-fold
# with H2O prior transformation. This can be achieved by mixing 5 μl of assembled products with
# 15 μl of H2O. Add 2 μl of the diluted assembled product to competent cells."
#
def _do_transformation():
#
# Combine plasmid and competent bacteria in a pcr_plate and shock
#
p.provision(inv["DH5a"], transform_tube, µl(50))
p.transfer(clone_plate.well(0), transform_tube, µl(3), dispense_speed="10:microliter/second")
assert clone_plate.well(0).volume == µl(54), clone_plate.well(0).volume
if 'control_pUC19' in options:
p.provision(inv["DH5a"], transctrl_tube, µl(50))
p.provision(inv["pUC19"], transctrl_tube, µl(1))
#
# Heatshock the bacteria to transform using a PCR machine
#
p.seal(transform_plate)
p.thermocycle(transform_plate,
[{"cycles": 1, "steps": [{"temperature": "4:celsius", "duration": "5:minute"}]},
{"cycles": 1, "steps": [{"temperature": "37:celsius", "duration": "30:minute"}]}],
volume=µl(50))
return
def _transfer_transformed_to_plates():
assert transform_tube.volume == µl(53), transform_tube.volume
p.unseal(transform_plate)
num_ab_plates = 4 # antibiotic places
#
# Transfer bacteria to a bigger tube for diluting
# Then spread onto 6-flat plates
# Generally you would spread 50-100ul of diluted bacteria
# Put more on ampicillin plates for more opportunities to get a colony
# I use a dilution series since it's unclear how much to plate
#
p.provision(inv["LB Miller"], transform_tube_L, µl(429))
#
# Add all IPTG and XGal to the master tube
# 4ul (1M) IPTG on each plate; 40ul XGal on each plate
#
p.transfer(IPTG_tube, transform_tube_L, µl(4*num_ab_plates))
if 'XGal' in options:
p.transfer(XGal_tube, transform_tube_L, µl(40*num_ab_plates))
#
# Add the transformed cells and mix (use new mix op in case of different pipette)
#
p.transfer(transform_tube, transform_tube_L, µl(50))
p.mix(transform_tube_L, volume=transform_tube_L.volume/2, repetitions=10)
assert transform_tube.volume == dead_volume['96-pcr'] == µl(3), transform_tube.volume
assert transform_tube_L.volume == µl(495), transform_tube_L.volume
#
# Spread an average of 60ul on each plate == 480ul total
#
for i in range(num_ab_plates):
p.spread(transform_tube_L, amp_6_flat.well(i), µl(51+i*6))
p.spread(transform_tube_L, noAB_6_flat.well(i), µl(51+i*6))
assert transform_tube_L.volume == dead_volume["micro-1.5"], transform_tube_L.volume
#
# Controls: include 2 ordinary pUC19-transformed plates as a control
#
if 'control_pUC19' in options:
num_ctrl = 2
assert num_ab_plates + num_ctrl <= 6
p.provision(inv["LB Miller"], transctrl_tube_L, µl(184)+dead_volume["micro-1.5"])
p.transfer(IPTG_tube, transctrl_tube_L, µl(4*num_ctrl))
if "XGal" in options: p.transfer(XGal_tube, transctrl_tube_L, µl(40*num_ctrl))
p.transfer(transctrl_tube, transctrl_tube_L, µl(48))
p.mix(transctrl_tube_L, volume=transctrl_tube_L.volume/2, repetitions=10)
for i in range(num_ctrl):
p.spread(transctrl_tube_L, amp_6_flat.well(num_ab_plates+i), µl(55+i*10))
p.spread(transctrl_tube_L, noAB_6_flat.well(num_ab_plates+i), µl(55+i*10))
assert transctrl_tube_L.volume == dead_volume["micro-1.5"], transctrl_tube_L.volume
assert IPTG_tube.volume == µl(808), IPTG_tube.volume
if "XGal" in options: assert XGal_tube.volume == µl(516), XGal_tube.volume
return
def do_transformation():
_do_transformation()
_transfer_transformed_to_plates()
# ------------------------------------------------------
# Measure growth in plates (photograph)
#
def measure_growth():
#
# Incubate and photograph 6-flat plates over 18 hours
# to see blue or white colonies
#
for flat_name, flat in [(expid("amp_6_flat"), amp_6_flat), (expid("noAB_6_flat"), noAB_6_flat)]:
for timepoint in [9,18]:
p.cover(flat)
p.incubate(flat, "warm_37", "9:hour")
p.uncover(flat)
p.image_plate(flat, mode="top", dataref=expid("{}_t{}".format(flat_name, timepoint)))
return
# ---------------------------------------------------------------
# Sanger sequencing, TURNED OFF
# Sequence to make sure assembly worked
# 500ng plasmid, 1 µl of a 10 µM stock primer
# "M13_F" : "rs17tcpqwqcaxe", # catalog; M13 Forward (-41); cold_20 (1ul = 100pmol)
# "M13_R" : "rs17tcph6e2qzh", # catalog; M13 Reverse (-48); cold_20 (1ul = 100pmol)
#
def do_sanger_seq():
seq_primers = [inv["M13_F"], inv["M13_R"]]
seq_wells = ["G1","G2"]
p.unseal(pcr_plate)
for primer_num, seq_well in [(0, seq_wells[0]),(1, seq_wells[1])]:
p.provision(seq_primers[primer_num], pcr_plate.wells([seq_well]), µl(1))
p.transfer(pcr_plate.wells(["A1"]), pcr_plate.wells(seq_wells), µl(5), mix_before=True, mix_vol=µl(10))
p.transfer(water_tube, pcr_plate.wells(seq_wells), µl(9))
p.mix(pcr_plate.wells(seq_wells), volume=µl(7.5), repetitions=10)
p.sangerseq(pcr_plate, pcr_plate.wells(seq_wells[0]).indices(), expid("seq1"))
p.sangerseq(pcr_plate, pcr_plate.wells(seq_wells[1]).indices(), expid("seq2"))
return
# ---------------------------------------------------------------
# Generate protocol
#
# Skip Gibson since I already did it
if 'gibson' in options: do_gibson_assembly()
do_transformation()
measure_growth()
if 'sanger' in options: do_sanger_seq()
# ---------------------------------------------------------------
# Output protocol
#
jprotocol = json.dumps(p.as_dict(), indent=2)
!echo '{jprotocol}' | transcriptic analyze
#print("nProtocol {}nn{}".format(experiment_name, jprotocol))
open("protocol_{}.json".format(experiment_name),'w').write(jprotocol)Inventory: IPTG/IPTG/IPTG/IPTG/IPTG/IPTG 832.0:microliter {}
Inventory: sfgfp_puc19_gibson_v3_clone/sfgfp_puc19_gibson_v3_clone/sfgfp_puc19_gibson_v3_clone/sfgfp_puc19_gibson_v3_clone/sfgfp_puc19_gibson_v3_clone 57.0:microliter {}
✓ Protocol analyzed
40 instructions
8 containers
Total Cost: $53.20
Workcell Time: $17.35
Reagents & Consumables: $35.86
Сбор колоний
Когда колонии растут на ампициллиновой пластине, я могу «собрать» отдельные колонии и посадить их на 96-пробирочный планшет. Для этого в автопротоколе есть специальная команда (autopick).
"""Pick colonies from plates and grow in amp media and check for fluorescence.
v2: try again with a new plate (no blue colonies)
v3: repeat with different emission and excitation wavelengths"""
p = Protocol()
options = {}
for k, v in list(options.items()):
if v is False: del options[k]
experiment_name = "sfgfp_puc19_gibson_pick_v3"
def plate_expid(val):
"""refer to the previous plating experiment's outputs"""
plate_exp = "sfgfp_puc19_gibson_plates_v4"
return "{}_{}".format(plate_exp, val)
# -----------------------------------------------------------------------
# Inventory
#
inv = {
# catalog
"water" : "rs17gmh5wafm5p", # catalog; Autoclaved MilliQ H2O; ambient
"LB Miller" : "rs17bafcbmyrmh", # catalog; LB Broth Miller; cold_4
"Amp 100mgml" : "rs17msfk8ujkca", # catalog; Ampicillin 100mg/ml; cold_20
"IPTG" : "ct18a2r5wn6tqz", # inventory; IPTG at 1M (conc semi-documented); cold_20
# plates from previous experiment, must be changed every new experiment
plate_expid("amp_6_flat") : "ct18snmr9avvg9", # inventory; Ampicillin plates with blue-white screening of pUC19
plate_expid("noAB_6_flat") : "ct18snmr9dxfw2", # inventory; no AB plates with blue-white screening of pUC19
}
# Tubes and plates
lb_amp_tubes = [p.ref("lb_amp_{}".format(i+1), cont_type="micro-2.0", storage="ambient", discard=True).well(0)
for i in range(4)]
lb_xab_tube = p.ref("lb_xab", cont_type="micro-2.0", storage="ambient", discard=True).well(0)
growth_plate = p.ref(expid("growth"), cont_type="96-flat", storage="cold_4", discard=False)
# My inventory
IPTG_tube = p.ref("IPTG", id=inv["IPTG"], cont_type="micro-1.5", storage="cold_20").well(0)
# ampicillin plate
amp_6_flat = Container(None, p.container_type('6-flat'))
p.refs[plate_expid("amp_6_flat")] = Ref(plate_expid("amp_6_flat"),
{"id":inv[plate_expid("amp_6_flat")], "store": {"where": 'cold_4'}}, amp_6_flat)
# Use a total of 50 wells
abs_wells = ["{}{}".format(row,col) for row in "BCDEF" for col in range(1,11)]
abs_wells_T = ["{}{}".format(row,col) for col in range(1,11) for row in "BCDEF"]
assert abs_wells[:3] == ["B1","B2","B3"] and abs_wells_T[:3] == ["B1","C1","D1"]
def prepare_growth_wells():
#
# To LB, add ampicillin at ~1/1000 concentration
# Mix slowly in case of overflow
#
p.provision(inv["LB Miller"], lb_xab_tube, µl(1913))
for lb_amp_tube in lb_amp_tubes:
p.provision(inv["Amp 100mgml"], lb_amp_tube, µl(2))
p.provision(inv["LB Miller"], lb_amp_tube, µl(1911))
p.mix(lb_amp_tube, volume=µl(800), repetitions=10)
#
# Add IPTG but save on X-Gal
# http://openwetware.org/images/f/f1/Dh5a_sub.pdf
# "If you are concerned about obtaining maximal levels of expression, add IPTG to a final concentration of 1 mM."
# 2ul of IPTG in 2000ul equals 1mM
#
p.transfer(IPTG_tube, [lb_xab_tube] + lb_amp_tubes, µl(2), one_tip=True)
#
# Distribute LB among wells, row D is control (no ampicillin)
#
cols = range(1,11)
row = "D" # control, no AB
cwells = ["{}{}".format(row,col) for col in cols]
assert set(cwells).issubset(set(abs_wells))
p.distribute(lb_xab_tube, growth_plate.wells(cwells), µl(190), allow_carryover=True)
rows = "BCEF"
for row, lb_amp_tube in zip(rows, lb_amp_tubes):
cwells = ["{}{}".format(row,col) for col in cols]
assert set(cwells).issubset(set(abs_wells))
p.distribute(lb_amp_tube, growth_plate.wells(cwells), µl(190), allow_carryover=True)
assert all(lb_amp_tube.volume == lb_xab_tube.volume == dead_volume['micro-2.0']
for lb_amp_tube in lb_amp_tubes)
return
def measure_growth_wells():
#
# Growth: absorbance and fluorescence over 24 hours
# Absorbance at 600nm: cell growth
# Absorbance at 615nm: X-gal, in theory
# Fluorescence at 485nm/510nm: sfGFP
# or 450nm/508nm (http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2695656/)
#
hr = 4
for t in range(0,24,hr):
if t > 0:
p.cover(growth_plate)
p.incubate(growth_plate, "warm_37", "{}:hour".format(hr), shaking=True)
p.uncover(growth_plate)
p.fluorescence(growth_plate, growth_plate.wells(abs_wells).indices(),
excitation="485:nanometer", emission="535:nanometer",
dataref=expid("fl2_{}".format(t)), flashes=25)
p.fluorescence(growth_plate, growth_plate.wells(abs_wells).indices(),
excitation="450:nanometer", emission="508:nanometer",
dataref=expid("fl1_{}".format(t)), flashes=25)
p.fluorescence(growth_plate, growth_plate.wells(abs_wells).indices(),
excitation="395:nanometer", emission="508:nanometer",
dataref=expid("fl0_{}".format(t)), flashes=25)
p.absorbance(growth_plate, growth_plate.wells(abs_wells).indices(),
wavelength="600:nanometer",
dataref=expid("abs_{}".format(t)), flashes=25)
return
# ---------------------------------------------------------------
# Protocol steps
#
prepare_growth_wells()
batch = 10
for i in range(5):
p.autopick(amp_6_flat.well(i), growth_plate.wells(abs_wells_T[i*batch:i*batch+batch]),
dataref=expid("autopick_{}".format(i)))
p.image_plate(amp_6_flat, mode="top", dataref=expid("autopicked_{}".format(i)))
measure_growth_wells()
# ---------------------------------------------------------------
# Output protocol
#
jprotocol = json.dumps(p.as_dict(), indent=2)
!echo '{jprotocol}' | transcriptic analyze
open("protocol_{}.json".format(experiment_name),'w').write(jprotocol)✓ Protocol analyzed 62 instructions 8 containers Total Cost: $66.38 Workcell Time: $57.59 Reagents & Consumables: $8.78
Результаты: сбор колоний
Сине–белый экран прекрасно показал, в основном, белые колонии на пластинах с антибиотиком (1-4) и только синие на пластинах без антибиотика (5-6). Это именно то, чего я ожидал, и я был рад это видеть, тем более, что использовал собственные IPTG и X-gal, которые я отправил в Transcriptic.

Сине–белый скрининг пластин с ампициллином (1-4) и без антибиотика (5-6)
Однако робот-сборщик колоний плохо работал с этими бело-синими колониями. Изображение ниже создано путём вычитания последовательных фотографий пластин после каждого раунда выбора пластины и увеличения контраста различий (в GraphicsMagick). Таким образом, я могу визуализировать, какие колонии были собраны (хотя и не идеально, поскольку собранные колонии не полностью удаляются).
Я также подписал изображение количеством колоний, собранных роботом Transcriptic. Предполагалось, что он соберёт максимум по 10 колоний из первых пяти пластин. Тем не менее, в целом было собрано несколько колоний, и это обычно это синие колонии. Роботу удалось только найти десять колоний на контрольной пластине с только синими колониями. Моя рабочая теория заключается в том, что робот, собирающий колонии, предпочтительно собирает синие колонии, поскольку они более контрастные.

Сине-белые скрининговые пластины с ампициллином (1-4) и без антибиотика (5-6), с указанием количества собранных колоний
Бело–голубой скрининг служил определённой цели. Он показал, что большинство колоний трансформируются правильно. По крайней мере, там происходит инсерция. Однако для лучшего сбора колоний я повторил эксперимент без X-gal.
Только с белыми колониями робот-сборщик успешно собрал по десять колоний из каждой из первых пяти пластин. Можно предположить, что в большинстве собранных колоний есть успешные инсерции.

Колонии, растущие на пластинах с ампициллином (1-4) и без антибиотика (5-6)
Результаты: трансформация с собранным продуктом
После выращивания 50 отобранных колоний на 96-пробирочном планшете в течение 20 часов я измеряю флуоресценцию, чтобы проверить экспрессию sfGFP. Для измерения флуоресценции и поглощения (и люминесценции, если хотите) Transcriptic использует ридер Tecan Infinite.
В теории, в любой колонии с ростом должна быть собранная плазмида, так как для роста ей нужна устойчивость к антибиотикам, и каждая собранная плазмида экспрессирует sfGFP. На самом деле есть много причин, почему это может быть не так, не в последнюю очередь из-за того, что вы можете потерять ген sfGFP из плазмиды без потери устойчивости к ампициллину. Бактерия, которая теряет ген sfGFP, имеет преимущество в отборе перед своими конкурентами, потому что она не тратит лишнюю энергию, а с учётом достаточного количества поколений роста, это обязательно произойдёт.
Я собираю данные поглощения (OD600) и флуоресценции каждые четыре часа в течение 20 часов (около 60 поколений).
for t in [0,4,8,12,16,20]:
abs_data = pd.read_csv("glow/sfgfp_puc19_gibson_pick_v3_abs_{}.csv".format(t), index_col="Well")
flr_data = pd.read_csv("glow/sfgfp_puc19_gibson_pick_v3_fl2_{}.csv".format(t), index_col="Well")
if t == 0:
new_data = abs_data.join(flr_data)
else:
new_data = new_data.join(abs_data, rsuffix='_{}'.format(t))
new_data = new_data.join(flr_data, rsuffix='_{}'.format(t))
new_data.columns = ["OD 600:nanometer_0", "Fluorescence_0"] + list(new_data.columns[2:])Размещаем на графике данные 20-го часа и следы предыдущих замеров. На самом деле мне интересны только последние данные, так как именно тогда должен наблюдаться пик флуоресценции.
svg = []
W, H = 800, 500
min_x, max_x = 0, 0.8
min_y, max_y = 0, 50000
def _toxy(x, y):
return W*(x-min_x)/(max_x-min_x), H-H*(y-min_y)/(max_y-min_y)
def _topt(x, y):
return ','.join(map(str,_toxy(x,y)))
ab_fls = [[row[0]] + [list(row[1])] for row in new_data.iterrows()]
# axes
svg.append('<g fill="#888" font-size="18" transform="translate(20,0),scale(.95)">')
svg.append('<text x="0" y="{}">OD600 →</text>'.format(H+20))
svg.append('<text x="0" y="0" transform="rotate(-90),translate(-{},-8)">Fluorescence →</text>'.format(H))
svg.append('<line x1="0" y1="{}" x2="{}" y2="{}" style="stroke:#888;stroke-width:2" />'.format(H,W,H))
svg.append('<line x1="0" y1="0" x2="0" y2="{}" style="stroke:#888;stroke-width:2" />'.format(H))
# glow filter
svg.append("""<filter id="glow" x="-200%" y="-200%" height="400%" width="400%">
<feColorMatrix type="matrix" values="0 0 0 0 0 255 0 0 0 0 0 0 0 0 0 0 0 0 1 0"/>
<feGaussianBlur stdDeviation="10" result="coloredBlur"/>
<feMerge><feMergeNode in="coloredBlur"/><feMergeNode in="SourceGraphic"/></feMerge>
</filter>""")
for n, (well, vals) in enumerate(ab_fls):
fill = "#444" if not well.startswith("D") else "#aaa"
gfilter = 'filter="url(#glow)"' if well in ["C3", "D1", "D3"] else ""
cx, cy = _toxy(*vals[-2:])
svg.append('''<g id="point{n:d}"><circle {gfilter:s} r="12" cx="{cx:f}" cy="{cy:f}" fill="{fill:s}" />
<text x="{cx:f}" y="{cy:f}" font-size="10" text-anchor="middle" fill="#fff"
alignment-baseline="middle">{txt:s}</text></g>
'''.format(n=n, cx=cx, cy=cy, fill=fill, txt=well, gfilter=gfilter))
pathd = 'M{} '.format(_topt(*vals[:2]))
pathd += ' '.join("L{}".format(_topt(*vals[i:i+2])) for i in range(2,len(vals),2))
svg.append('''<path d="{pathd:}" stroke="#ccc" stroke-width=".2"
fill="none" id="path{n:d}"/>'''.format(pathd=pathd, n=n))
svg.append("</g>") # entire chart group
show_svg(''.join(svg), w=W, h=H)
Флуоресценция и OD600: колонии с ампициллином чёрные, контрольные колонии без ампициллина серые. Зелёным подсвечены колонии, где я подтвердил правильность последовательности белка sfGFP
Запускаем miniprep, чтобы извлечь плазмидную ДНК, затем последовательность Сэнгера с помощью праймеров М13. К сожалению, по какой-то причине miniprep в настоящее время доступен только через веб-протокол Transcriptic, а не через автопротокол. Я секвенирую три пробирки с самыми высокими показаниями флуоресценции (C1, D1, D3) и три других (B1, B3, E1), выравнивая последовательности по sfGFP с помощью muscle.
В пробирках C1, D3 и D3 идеальное соответствие моей исходной последовательности sfGFP, в то время как в B1, B3 и E1 грубые мутации или выравнивание просто терпит неудачу.
Три светящиеся колонии
Результаты хорошие, хотя некоторые аспекты удивительны. Например, ридер флуоресценции без видимой причины начинает работу с временной отметки 0 с очень высоких значений (40 000 блоков). К 20-му часу он успокоился до более разумного паттерна с чёткой корреляцией между OD600 и флуоресценцией (предполагаю, из-за небольшого перекрытия в спектрах), плюс некоторые выбросы с высокой флуоресценцией. Удивительно, но это может быть один, три или, возможно, 11-15 выбросов.
Некоторые из пробирок с высокими показателями флуоресценции находятся в контрольных пробирках (т. е. без ампициллина, окрашены серым), что удивительно, поскольку в этих пробирках нет давления отбора, поэтому можно ожидать потери плазмиды).
Основываясь на данных флуоресценции и результатах секвенирования, представляется, что только три из 50 колоний производят sfGFP и флуоресцируют. Это не так много, как я ожидал. Однако, поскольку прошло три отдельные стадии роста (на пластине, в пробирке, для miniprep), к этой стадии клетки претерпели около 200 поколений роста, поэтому было довольно много возможностей для возникновения мутаций.
Должны быть способы сделать процесс более эффективным, тем более что я далеко не эксперт по этим протоколам. Тем не менее, мы успешно произвели трансформированные клетки с экспрессией сконструированного GFP, используя только код Python!
Часть третья: выводы
Цена
В зависимости от того, как измерять, стоимость этого эксперимента составила около $360, не считая денег на отладку:
- $70 на синтез ДНК
- $32 на PCR и добавление фланкирующих последовательностей к инсерции
- $31 на вырезание плазмид
- $32 на сборку Гибсона
- $53 на трансформацию
- $67 на сбор колоний
- $75 за 3 miniprep'ов и секвенирование
Думаю, что стоимость можно снизить до $250-300 с некоторыми доработками. Например, роботизированный сбор 50 колоний стоит подозрительно дорого и, вероятно, от него можно отказаться.
По моему опыту, эта цена кажется высокой для некоторых (молекулярных биологов) и низкой для других (людей из IT). Поскольку Transcriptic в основном просто взимает плату за реагенты по прейскуранту, основная разница в затратах заключается в труде. Робот и так обходится довольно дёшево в час, и он не прочь встать посреди ночи, чтобы сфотографировать пластину. После того, как протоколы утверждены, трудно представить, что даже аспирант обойдётся дешевле, особенно если учитывать альтернативные издержки.
Для ясности, я говорю только о замене рутинных протоколов. Конечно, разработка передовых протоколов по-прежнему будет осуществляться квалифицированными молекулярными биологами, но многие захватывающие области науки используют скучные рутинные протоколы. До недавнего времени многие лаборатории производили свои собственные олигонуклеотиды, но теперь мало кто беспокоится об этом: это просто не стоит ничьего времени, даже аспирантов, когда IDT отправит их вам в течение нескольких дней.
Роботизированные лаборатории: плюсы и минусы
Очевидно, я очень верю в будущее роботизированных лабораторий. Есть некоторые действительно забавные и полезные вещи в проведении экспериментов с роботами, особенно если вы в первую очередь занимаетесь вычислениями и у вас аллергия на латексные перчатки и ручной труд:
- Воспроизводимость! Вероятно, это главное преимущество. Воспроизводимость включает в себя согласованность роботов и возможность публиковать свой протокол в формате autoprotocol, а не неудобной английской прозой.
- Масштабируемость. Вы можете повторить мой эксперимент 100 раз с разными параметрами, без особых запредельных усилий.
- Произвольно сложные протоколы, например, тачдаун PCR. Это может показаться незначительным или даже контрпродуктивным, но если протокол выполняется сотни или тысячи раз разными лабораториями, почему бы немного не оптимизировать его? Или даже использовать статистику/машинное обучение для улучшения протокола с течением времени? Меня сводит с ума, когда встречаются протоколы, которые предполагается использовать десятки тысяч раз, а они рекомендуют выполнять операцию «в течение 2-3 минут». Сколько именно?
- Тонкая настройка. Вы можете повторить эксперимент после изменения только одной незначительной детали. Человеку очень сложно сделать такое, при прочих равных.
- Виртуальность. Запускайте эксперименты и контролируйте результат на расстоянии.
- Выразительность. Можно использовать синтаксис программирования для кодирования повторяющихся шагов или логики ветвления. Например, если вы хотите дозировать от 1 до 96 мкл реагента и (96−x)мкл воды в 96-пробирочный планшет, это можно кратко записать.
- Машиночитаемые данные. Данные с результатами почти всегда возвращаются в формате csv или другом формате, пригодном для машинной обработки.
- Абстракция. В идеале вы можете запустить весь протокол независимо от используемых реагентов или стиля клонирования и что-то заменить в случае необходимости, если оно работает лучше.
Конечно, есть и некоторые недостатки, тем более, что инструменты только начали развиваться. Если сравнить с интернетом, то мы в районе 1994 года:
- Транспортировка образцов к Transcriptic и назад — тяжёлая работа. Я не уверен, как это лучше организовать, но чем больше вы можете сделать в облачной лаборатории, тем меньше нужно транспортировать. Отчасти поэтому синтетическая биология хорошо подходит для облачных лабораторий, скажем, для диагностики с человеческими образцами.
- Удалённая отладка протоколов сложна и может быть дорогостоящей — особенно попытки различить свои ошибки и баги Transcriptic.
- Есть много экспериментов, которые вы просто не можете сделать. На момент написания статьи Transcriptic поддерживает только бактериальные эксперименты (без дрожжей, без клеток млекопитающих, хотя они появятся в будущем).
- Для многих лабораторий может быть дороже использовать облачную лабораторию, чем просто взять аспиранта для выполнения работы (предельные затраты в час: ~$0). Это зависит от того, нужны лаборатории руки аспиранта или его интеллект.
- Transcriptic ещё не проводит экспериментов в выходные. Их можно понять, но такое бывает неудобно, даже если у вас небольшой проект.
Софт для изготовления белков
Хотя здесь довольно много кода и довольно много отладки, я думаю, что возможно создать какое-то программное обеспечение, которое принимает на вход последовательность белков, а на выходе создаёт бактерии с экспрессией этого белка.
Чтобы это сработало, должно произойти несколько вещей:
- Истинная интеграция Twist/IDT/Gen9 с Transcriptic (вероятно, она будет происходить медленно из-за низкого спроса в настоящее время).
- Очень надёжные версии протоколов, которые я описал выше, чтобы учесть различия в составе последовательности белков, длине, вторичной структуре и т. д.
- Замена различных проприетарных инструментов (генератор протоколов Гибсона от NEB, оптимизатор кодонов от IDT) на эквиваленты с открытым исходным кодом (например, primer3).
Во многих приложениях вы также хотите очистить свой белок (через колонку) или, возможно, просто заставить бактерии секретировать его. Предположим, что вскоре мы сможем сделать это в облачной лаборатории или что сможем проводить эксперименты in vivo (т. е. внутри бактериальной клетки).
Есть много возможностей для того, чтобы протокол на самом деле работал лучше человека, например: дизайн промоторов и RBS для оптимизации экспрессии, специфичной для вашей последовательности; статистика вероятности успеха эксперимента на основе сопоставимых экспериментов; автоматизированный анализ гелей.
Зачем всё это?
После всего этого может быть не совсем ясно, зачем создавать такой белок. Вот некоторые идеи:
- Сделать датчик для обнаружения чего-то опасного/нездорового/вкусного, как глютен.
- Сделать вакцину с идентификацией пептидов, уникальных для патогена, и хорошенько подумать.
- Оценить связи белка in vivo с помощью split-GFP или схожих подходов.
- Сделать scFv в качестве датчика для сальмонеллы. Эти scFvs действуют как мини-антитела с фолдингом внутри бактерии.
- Сделать BiTE для лечения конкретного вида рака, который вы только что секвенировали (это может быть сложнее, чем кажется).
- Сделать местную вакцину, которая входит в организм через волосяные фолликулы (не рекомендую пробовать это дома).
- Мутагенизировать свой белок сотней разных способов и посмотреть, что получится. Затем масштабировать до 1000 или 10 000 мутаций? Может, охарактеризовать мутации GFP?
Для новых идей того, что возможно при конструировании белков, посмотрите на сотни проектов iGEM.
В конце хочу выразить благодарность Бену Майлзу из Transcriptic за помощь в завершении этого проекта.
Автор: m1rko