Всем привет.
Видел несколько дашбордов по COVID-19, но не нашёл пока главного — прогноза времени спада эпидемии. Поэтому написал небольшой скрипт на Python. Он забирает данные из таблиц ВОЗ на Github'е, раскладывает по странам, строит линии тренда. И по ним делает прогнозы — когда в каждой стране из ТОП 20 по количеству заболевших COVID-19 можно ожидать спада заражений. Писал на скорую руку, так что не обессудьте. Если интересуют результаты — добро пожаловать под cut.
Немного о дашбордах
На первых порах для того, чтобы оценить сроки эпидемии я пользовался общедоступными дашбордами. Одним из первых стал сайт Центра системных наук и инженерии Университета Джона Хопкинса (Center for Systems Science and Engineering of Johns Hopkins University).
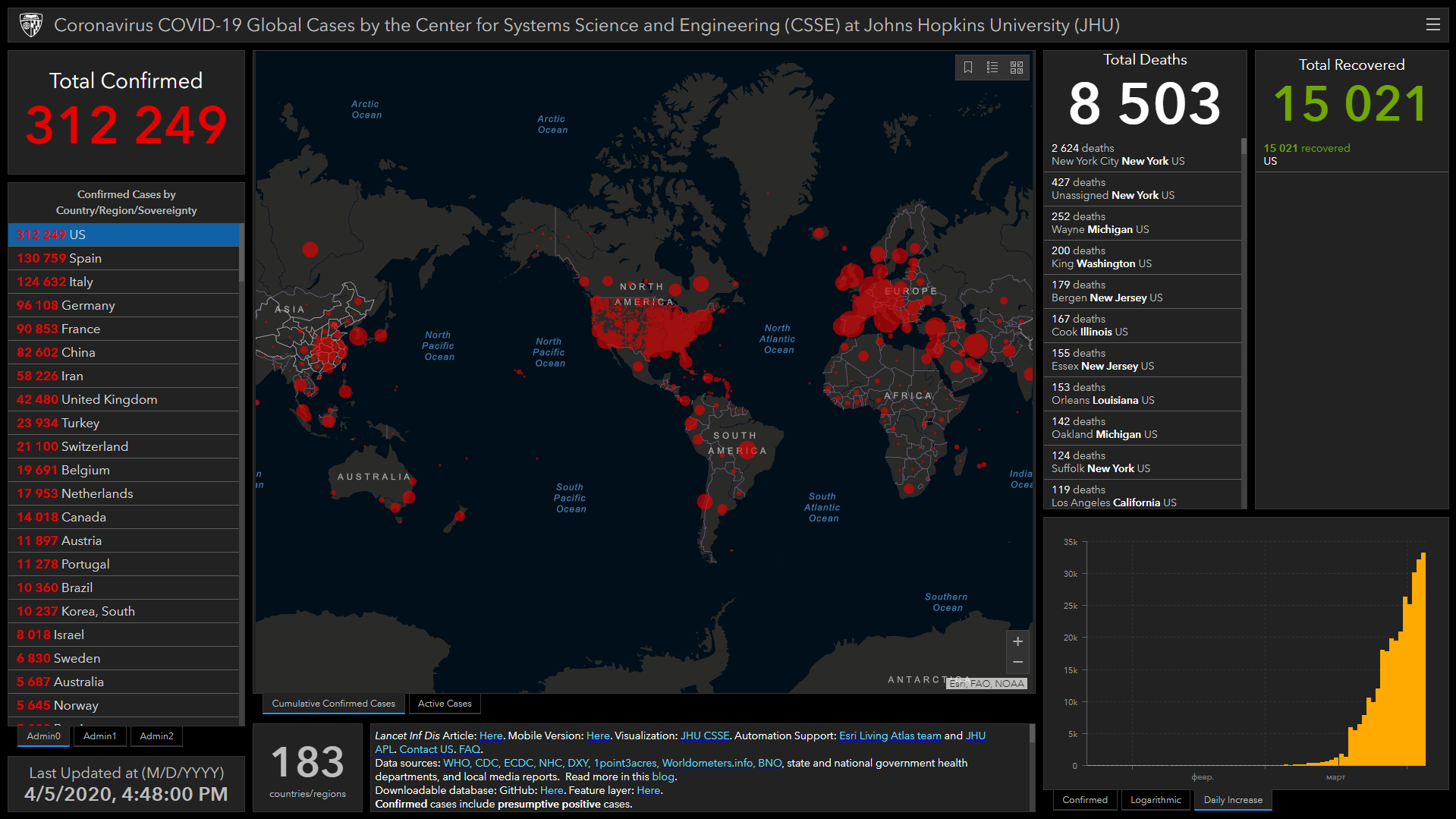
Он довольно симпатичен своей зловещей простотой. До недавнего времени не позволял строить динамику приращения заражений по дням, но исправился. И любопытен тем, что позволяет видеть распространение пандемии на карте мира. Учитывая черно-красную гамму дашборда, долго не него смотреть всё же не рекомендуется. Думаю, это может быть чревато возникновением приступов тревожности, переходящих в различные формы паники.
Мне для того, чтобы держать руку на пульсе больше понравилась страничка про COVID-19 на ресурсе Worldomert. На ней информация по странам представлена в виде таблицы:
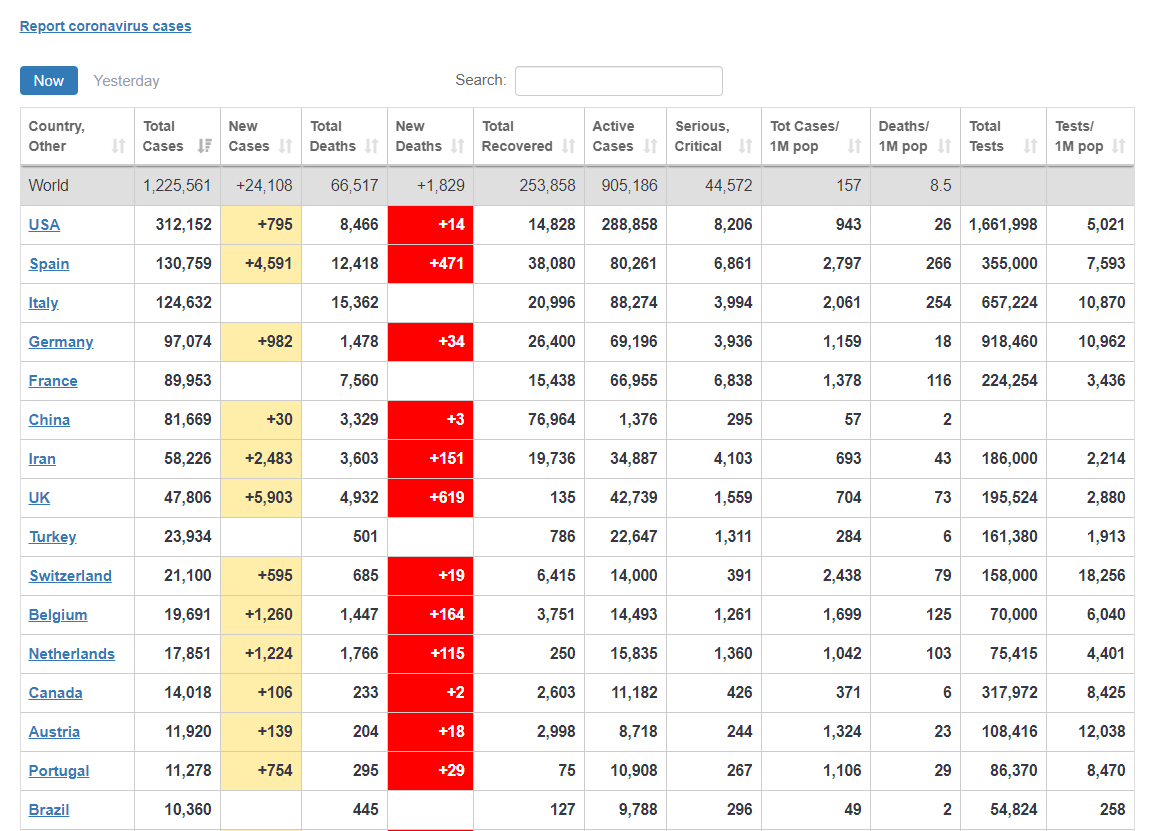
При клике на нужную страну мы проваливаемся в более подробную информацию о ней, включая дневные и накопительные кривые по зараженным/выздоровевшим/погибшим.
Есть и другие дашборды. Например, для углубленной информации по нашей стране можно просто в строке поиска Яндекса вбить «COVID-19» и Вы попадёте на такой:
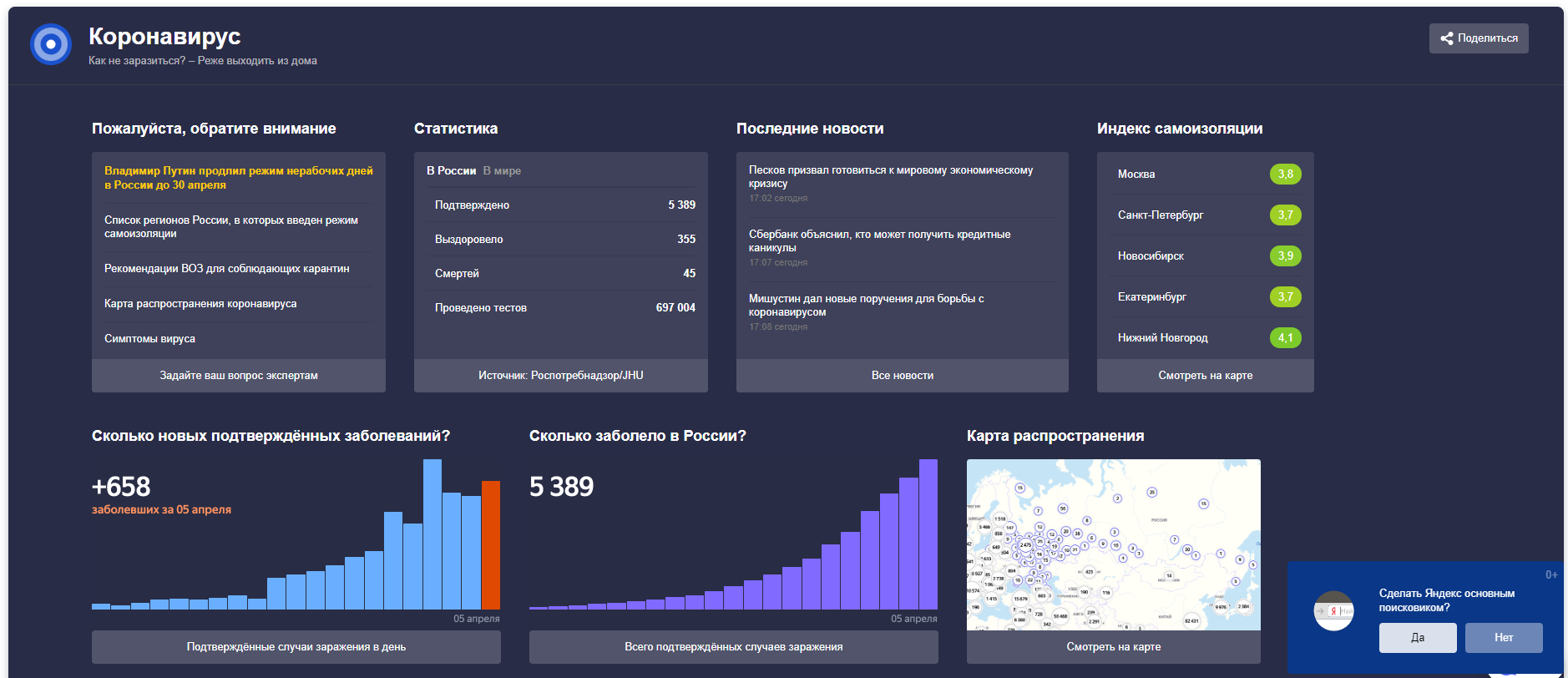
Ну, пожалуй, на этом и остановимся. Ни на этих трёх дашбордах, ни на нескольких других, мне не удалось найти информации с прогнозами, когда наконец пандемия пойдет на спад.
Немного о графиках
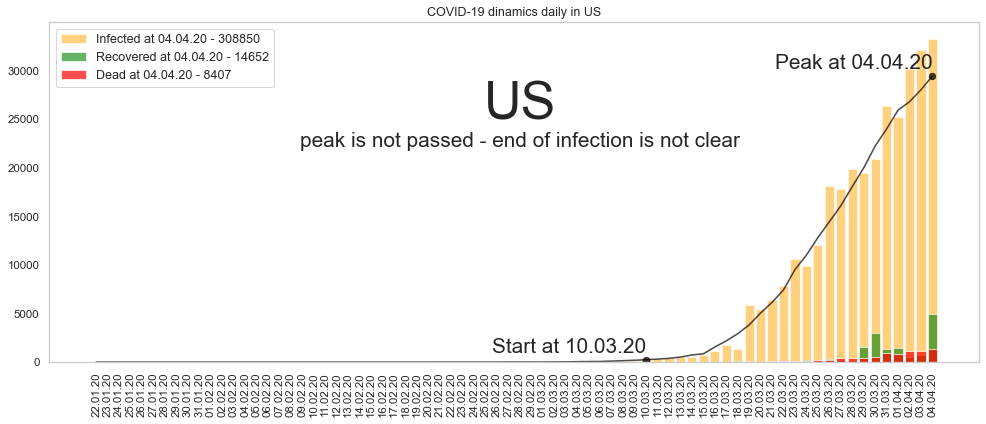
В настоящее время заражение только набирает обороты и наибольший интерес представляют графики с ежедневным приростом количества заболевших. Вот пример для США:
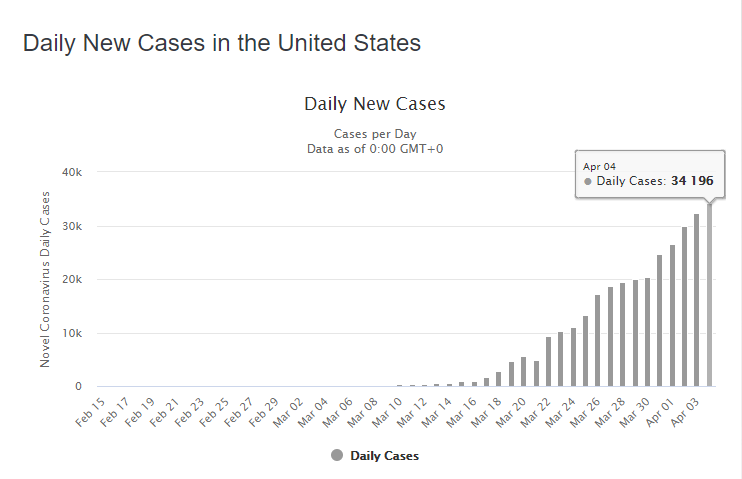
Видно, что количество заразившихся растёт каждый день. На подобных графиках речь, конечно же, идёт не о вообще всех заразившихся (их точное количество определить не представляется возможным), а только о подтверждённых случаях. Но для краткости здесь и в дальнейшем будем этих подтверждённых заразившихся называть просто «заразившимися».
Как выглядит укрощённая пандемия? Образцом здесь может послужить график Южной Кореи:
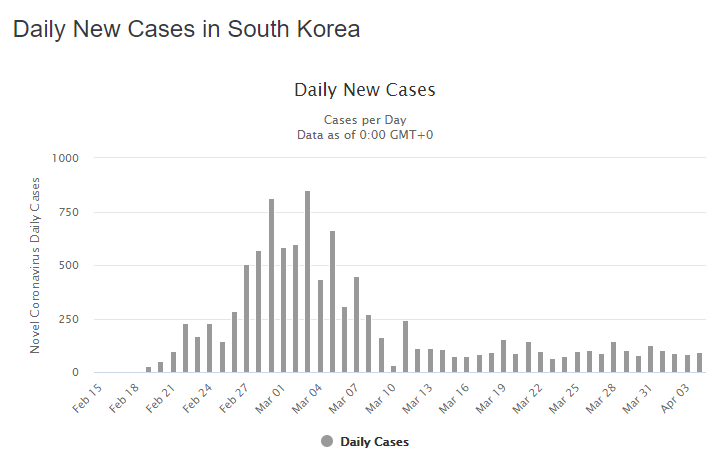
Видно, что ежедневное приращение заразившихся сначала росло, потом достигло пика, перешло к снижению и зафиксировалось на неком постоянном уровне (порядка 100 человек в день). Это ещё не полная победа над вирусом, но значительный успех. Это позволило стране запустить экономику (хотя, говорят, корейцы умудрились её не тормозить) и вернуться к более менее нормальной жизни.
Можно предположить, что ситуация и в других странах будет развиваться сходным образом. То есть рост на первом этапе сменит стабилизация, а потом количество ежедневно заболевших пойдёт на спад. В результате получится колоколообразная кривая. Давайте поищем пики кривых наиболее «заразившихся» стран.
Загружаем и обрабатываем данные
Данные по распространению коронавируса обновляются на постоянной основе здесь. В исходных таблицах содержатся накопительные кривые, их можно забрать и предварительно обработать небольшим скриптом.
import pandas as pd
import ipywidgets as widgets
from ipywidgets import interact
import matplotlib.pyplot as plt
pd.set_option('display.max_columns', None)
# Загружаем данные
url = 'https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/'
confirmed = pd.read_csv(url + 'time_series_covid19_confirmed_global.csv', sep = ',')
deaths = pd.read_csv(url + 'time_series_covid19_deaths_global.csv', sep = ',')
recovered = pd.read_csv(url + 'time_series_covid19_recovered_global.csv', sep = ',')
# Пишем даты по-человечески в формате 'dd.mm.yy'
new_cols = list(confirmed.columns[:4]) + list(confirmed.columns[4:].map(lambda x: '{0:02d}.{1:02d}.{2:d}'.format(int(x.split(sep='/')[1]), int(x.split(sep='/')[0]), int(x.split(sep='/')[2]))))
confirmed.columns = new_cols
recovered.columns = new_cols
deaths.columns = new_cols
# Формируем таблицы с ежедневными приращениями
confirmed_daily = confirmed.copy()
confirmed_daily.iloc[:,4:] = confirmed_daily.iloc[:,4:].diff(axis=1)
deaths_daily = deaths.copy()
deaths_daily.iloc[:,4:] = deaths_daily.iloc[:,4:].diff(axis=1)
recovered_daily = recovered.copy()
recovered_daily.iloc[:,4:] = recovered_daily.iloc[:,4:].diff(axis=1)
# Формируем таблицу со сглаженными ежедневными приращениями заболевших
smooth_conf_daily = confirmed_daily.copy()
smooth_conf_daily.iloc[:,4:] = smooth_conf_daily.iloc[:,4:].rolling(window=8, min_periods=2, center=True, axis=1).mean()
smooth_conf_daily.iloc[:,4:] = smooth_conf_daily.iloc[:,4:].round(1)
# Определяем последнюю дату, на которую есть данные
last_date = confirmed.columns[-1]
# Определяем 20 стран с максимальным количеством подтвержденных случаев заражения
confirmed_top = confirmed.iloc[:, [1, -1]].groupby('Country/Region').sum().sort_values(last_date, ascending = False).head(20)
countries = list(confirmed_top.index)
# Определяем, какую долю зараженных дают эти 20 стран
conf_tot_ratio = confirmed_top.sum() / confirmed.iloc[:, 4:].sum()[-1]
# Добавляем к списку Россию
countries.append('Russia')
# Формируем легенду с колчеством подтвержденных случаев зараженных, вылечившихся и умерших
l1 = 'Infected at ' + last_date + ' - ' + str(confirmed.iloc[:, 4:].sum()[-1])
l2 = 'Recovered at ' + last_date + ' - ' + str(recovered.iloc[:, 4:].sum()[-1])
l3 = 'Dead at ' + last_date + ' - ' + str(deaths.iloc[:, 4:].sum()[-1])
# Выводим на графике три суммарные кривые
fig, ax = plt.subplots(figsize = [16,6])
ax.plot(confirmed.iloc[:, 4:].sum(), '-', alpha = 0.6, color = 'orange', label = l1)
ax.plot(recovered.iloc[:, 4:].sum(), '-', alpha = 0.6, color = 'green', label = l2)
ax.plot(deaths.iloc[:, 4:].sum(), '-', alpha = 0.6, color = 'red', label = l3)
ax.legend(loc = 'upper left', prop=dict(size=12))
ax.xaxis.grid(which='minor')
ax.yaxis.grid()
ax.tick_params(axis = 'x', labelrotation = 90)
plt.title('COVID-19 in all countries. Top 20 countries consists {:.2%} of total confirmed infected cases.'.format(conf_tot_ratio[0]))
plt.show()
Таким образом, можно получить такие же кривые, как и на дашбордах:

В скрипте есть немного лишнего, что не нужно для вывода графика, но понадобится на следующих этапах. Например, я сразу сформировал список из 20 стран с максимальным количеством зараженных на сегодняшний день. К слову — это 90% от числа всех заразившихся.
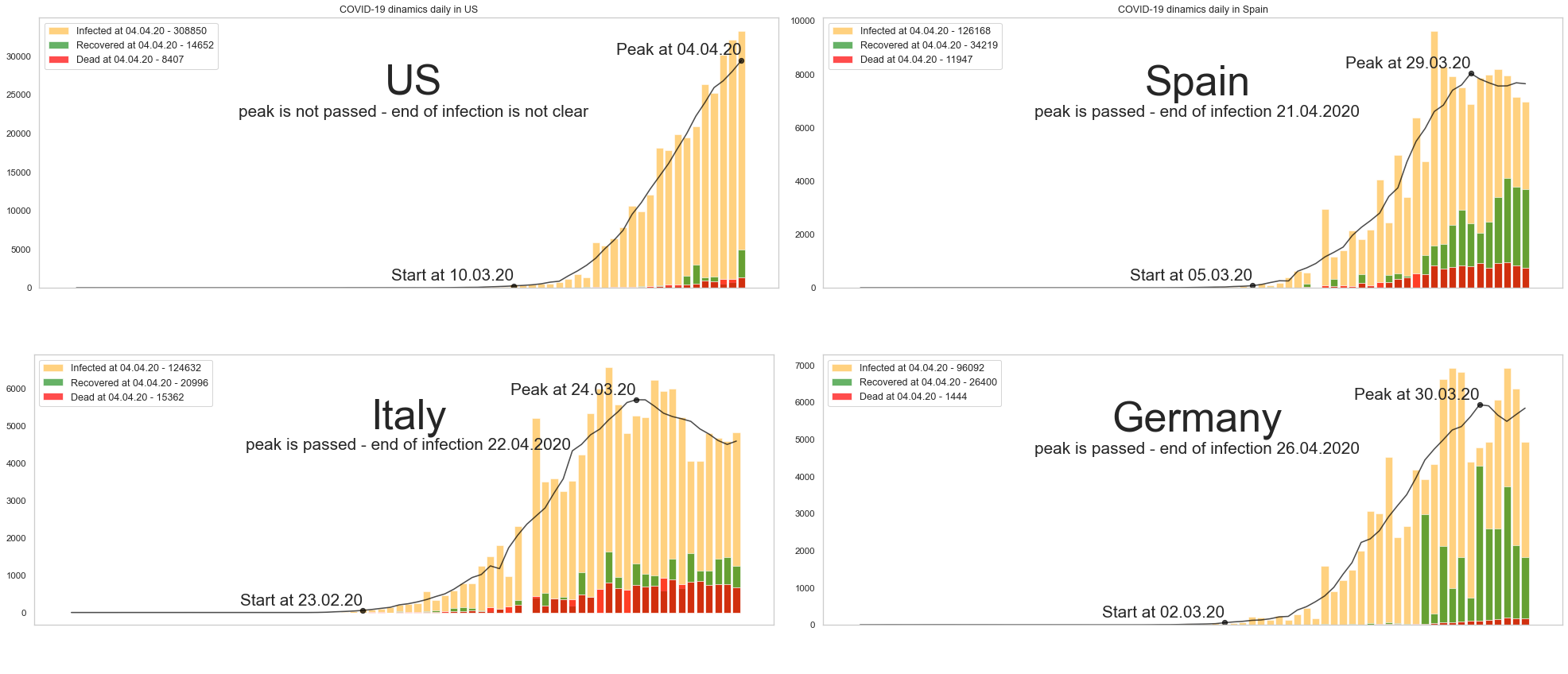
Идём дальше. Цифры дневного прироста заболевших довольно сильно «зашумлены». Поэтому график неплохо бы сгладить. Добавим на дневные графики линию тренда (я использовал отцентрированное скользящее среднее). Получается как-то так:

Тренд показан черным. Это довольно гладкая кривая с единственным пиком. Осталось находить начало всплеска заболевания (красная стрелка) и пик заболеваемости (зеленая). С пиком всё просто — это максимум на сглаженных данных. При этом, если максимум приходится на крайнюю справа точку графика, значит, пик ещё не пройден. Если максимум остался левее — скорее всего пик остался в прошлом.
Начало всплеска можно находить по-разному. Предположим для простоты, что это момент времени, когда количество зараженных за день начинает превышать 1% от максимального количества зараженных за день.
График при этом является довольно симметричным. То есть продолжительность от начала эпидемии до пика примерно равно продолжительности от пика до спада. Таким образом, если мы найдём количество дней от начала до пика, то примерно столько же дней нужно будет подождать до момента, когда эпидемия спадёт.
Заложим всю эту логику в следующем скрипте.
from datetime import timedelta, datetime
# Формируем сводную таблицу по 20 странам + Россия.
confirmed_top = confirmed_top.rename(columns={str(last_date): 'total_confirmed'})
dates = [i for i in confirmed.columns[4:]]
for country in countries:
# Находим для каждой страны общее число заразившихся, прирост их числа на текущую дату, уровень смертности
confirmed_top.loc[country, 'total_confirmed'] = confirmed.loc[confirmed['Country/Region'] == country,:].sum()[4:][-1]
confirmed_top.loc[country, 'last_day_conf'] = confirmed.loc[confirmed['Country/Region'] == country,:].sum()[-1] - confirmed.loc[confirmed['Country/Region'] == country,:].sum()[-2]
confirmed_top.loc[country, 'mortality, %'] = round(deaths.loc[deaths['Country/Region'] == country,:].sum()[4:][-1]/confirmed.loc[confirmed['Country/Region'] == country,:].sum()[4:][-1]*100, 1)
# Пиком эпидемии считаем точку максимума линии тренда.
smoothed_conf_max = round(smooth_conf_daily[smooth_conf_daily['Country/Region'] == country].iloc[:,4:].sum().max(), 2)
peak_date = smooth_conf_daily[smooth_conf_daily['Country/Region'] == country].iloc[:,4:].sum().idxmax()
peak_day = dates.index(peak_date)
# За время старта эпидемии принимаем дату, на кторую колчиество заболевших превышает 1% от максимума дневных заражений
start_day = (smooth_conf_daily[smooth_conf_daily['Country/Region'] == country].iloc[:,4:].sum() < smoothed_conf_max/100).sum()
start_date = dates[start_day-1]
# Записываем результаты в сводную таблицу
confirmed_top.loc[country, 'trend_max'] = smoothed_conf_max
confirmed_top.loc[country, 'start_date'] = start_date
confirmed_top.loc[country, 'peak_date'] = peak_date
confirmed_top.loc[country, 'peak_passed'] = round(smooth_conf_daily.loc[smooth_conf_daily['Country/Region'] == country, last_date].sum(), 2) != smoothed_conf_max
confirmed_top.loc[country, 'days_to_peak'] = peak_day - start_day
# Если пик пройдет, вычисляем время завершения эпидемии
if confirmed_top.loc[country, 'peak_passed']:
confirmed_top.loc[country, 'end_date'] = (datetime.strptime(confirmed_top.loc[country, 'peak_date']+'20', "%d.%m.%Y").date() + timedelta(confirmed_top.loc[country, 'days_to_peak'])).strftime('%d.%m.%Y')
# Фиксим данные по Китаю
confirmed_top.loc['China', 'start_date'] = '22.01.20'
confirmed_top
На выходе получим вот такую таблицу по топ 20 стран + Россия (она на момент написания статьи в двадцатку не входила, и, надеюсь, не войдёт).

В таблице следующие столбцы:
total_confirmed — общее число заразившихся в стране;
last_day_conf — число заразившихся за последний день;
mortality, % — уровень смертности (кол-во погибших/кол-во заразившихся);
trend_max — максимум тренда;
start_date — дата начала эпидемии в стране;
peak_date — дата достижения пика;
peak_passed — флажок пика (если True — пик пройден);
days_to_peak — сколько дней прошло от начала до пика;
end_date — дата окончания эпидемии.
Как видим, пик заболеваемости пройден в 12 странах из 20. Причём в среднем от начала массовых заражений до пика проходит в среднем 25 дней:

Согласно вышеизложенной логике, к концу апреля практически во всех рассматриваемых странах ситуация должна будет нормализоваться. Как будет на самом деле, покажет лишь время.
Есть ещё вот такой скриптик, который позволяет выводить графики по каждой из стран.
@interact
def plot_by_country(country = countries):
# Формируем надписи легенды
l1 = 'Infected at ' + last_date + ' - ' + str(confirmed.loc[confirmed['Country/Region'] == country,:].sum()[-1])
l2 = 'Recovered at ' + last_date + ' - ' + str(recovered.loc[recovered['Country/Region'] == country,:].sum()[-1])
l3 = 'Dead at ' + last_date + ' - ' + str(deaths.loc[deaths['Country/Region'] == country,:].sum()[-1])
# Формируем временный датафрейм с данными по выбранной стране
df = pd.DataFrame(confirmed_daily.loc[confirmed_daily['Country/Region'] == country,:].sum()[4:])
df.columns = ['confirmed_daily']
df['recovered_daily'] = recovered_daily.loc[recovered_daily['Country/Region'] == country,:].sum()[4:]
df['deaths_daily'] = deaths_daily.loc[deaths_daily['Country/Region'] == country,:].sum()[4:]
df['deaths_daily'] = deaths_daily.loc[deaths_daily['Country/Region'] == country,:].sum()[4:]
df['smooth_conf_daily'] = smooth_conf_daily.loc[smooth_conf_daily['Country/Region'] == country,:].sum()[4:]
# Рисуем график
fig, ax = plt.subplots(figsize = [16,6], nrows = 1)
plt.title('COVID-19 dinamics daily in ' + country)
# Наносим ежедные цифры по приросту заразившихся, выздоровевших и погибших
ax.bar(df.index, df.confirmed_daily, alpha = 0.5, color = 'orange', label = l1)
ax.bar(df.index, df.recovered_daily, alpha = 0.6, color = 'green', label = l2)
ax.bar(df.index, df.deaths_daily, alpha = 0.7, color = 'red', label = l3)
ax.plot(df.index, df.smooth_conf_daily, alpha = 0.7, color = 'black')
# Наносим даты начала и пика.
start_date = confirmed_top[confirmed_top.index == country].start_date.iloc[0]
start_point = smooth_conf_daily.loc[smooth_conf_daily['Country/Region'] == country, start_date].sum()
ax.plot_date(start_date, start_point, 'o', alpha = 0.7, color = 'black')
shift = confirmed_top.loc[confirmed_top.index == country, 'trend_max'].iloc[0]/40
plt.text(start_date, start_point + shift, 'Start at ' + start_date, ha ='right', fontsize = 20)
peak_date = confirmed_top[confirmed_top.index == country].peak_date.iloc[0]
peak_point = smooth_conf_daily.loc[smooth_conf_daily['Country/Region'] == country, peak_date].sum()
ax.plot_date(peak_date, peak_point, 'o', alpha = 0.7, color = 'black')
plt.text(peak_date, peak_point + shift, 'Peak at ' + peak_date, ha ='right', fontsize = 20)
ax.xaxis.grid(False)
ax.yaxis.grid(False)
ax.tick_params(axis = 'x', labelrotation = 90)
ax.legend(loc = 'upper left', prop=dict(size=12))
# Выводим название страны крупно и прогнозную дата спада эпидемии.
max_pos = max(df['confirmed_daily'].max(), df['recovered_daily'].max())
if confirmed_top[confirmed_top.index == country].peak_passed.iloc[0]:
estimation = 'peak is passed - end of infection ' + confirmed_top[confirmed_top.index == country].end_date.iloc[0]
else:
estimation = 'peak is not passed - end of infection is not clear'
plt.text(df.index[len(df.index)//2], 3*max_pos//4, country, ha ='center', fontsize = 50)
plt.text(df.index[len(df.index)//2], 2*max_pos//3, estimation, ha ='center', fontsize = 20)
#plt.savefig(country + '.png', bbox_inches='tight', dpi = 75)
plt.show()
Вот текущая ситуация по России:
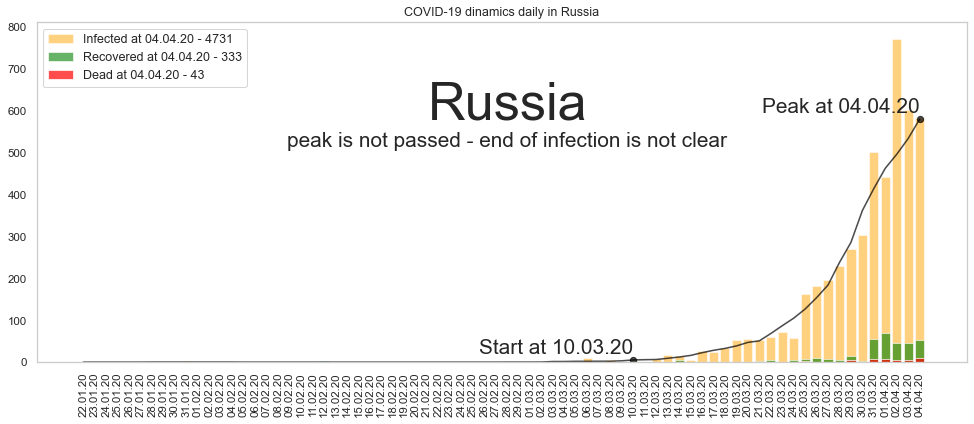
К сожалению, пик пока ещё не пройден, поэтому трудно делать прогнозы относительно сроков снижения заболеваемости. Но учитывая, что вспышка длится на текущий момент 24 дня, можно ожидать, что в течение недели мы увидим пик и заболеваемость начнёт снижаться. Рискну предположить, что к началу мая ситуация нормализуется (я всегда был оптимистом, надо сказать).
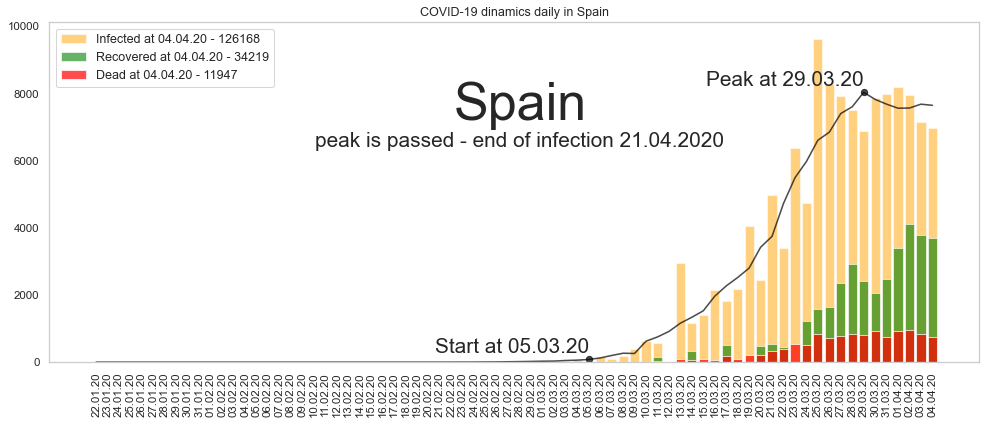
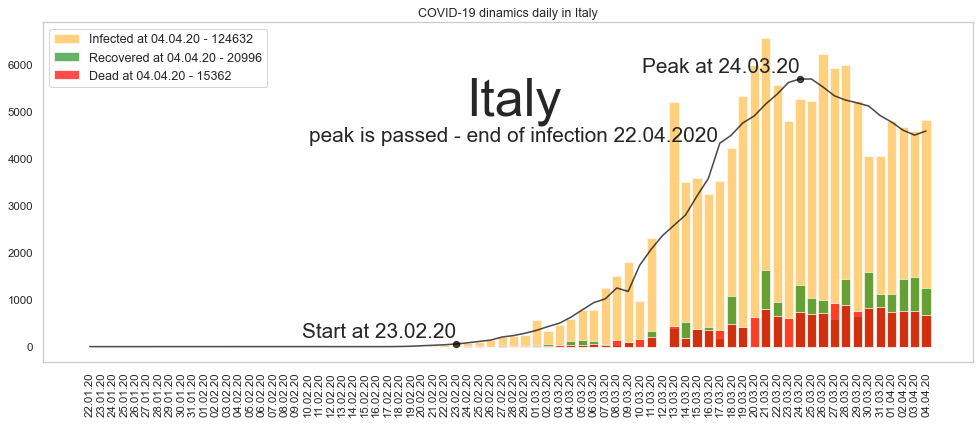
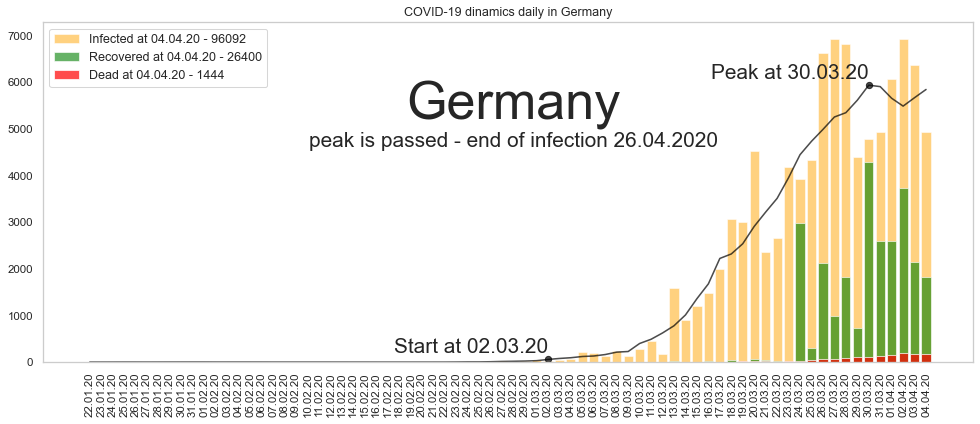
Большое спасибо за то, что дочитали до конца. Будьте здоровы и пребудет с нами сила. Ниже графики по остальным странам из двадцатки в порядке убывания по числу заразившихся.
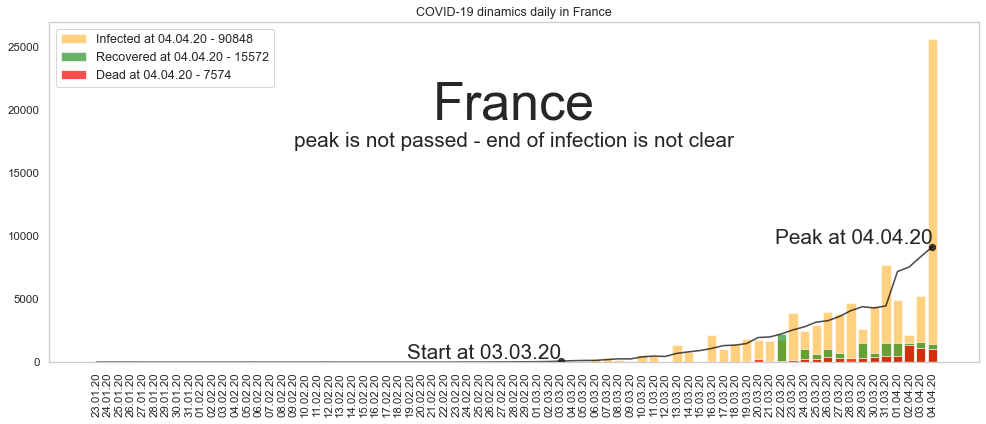
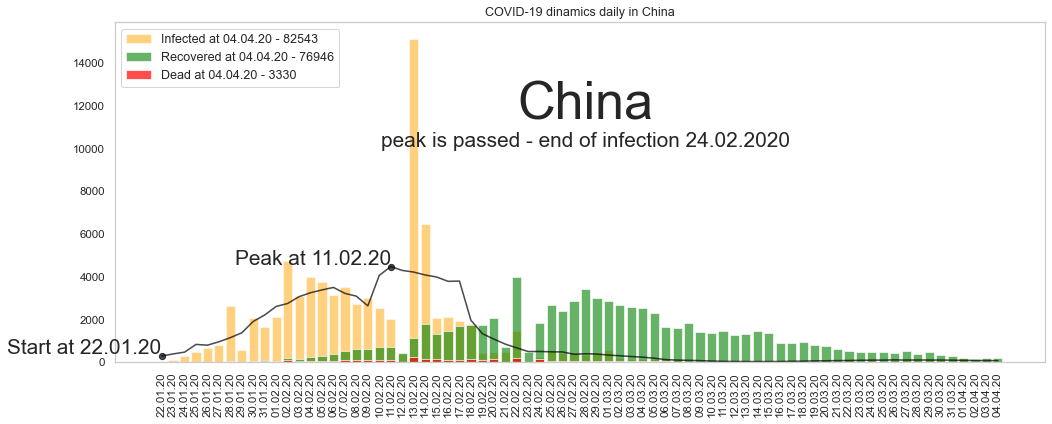
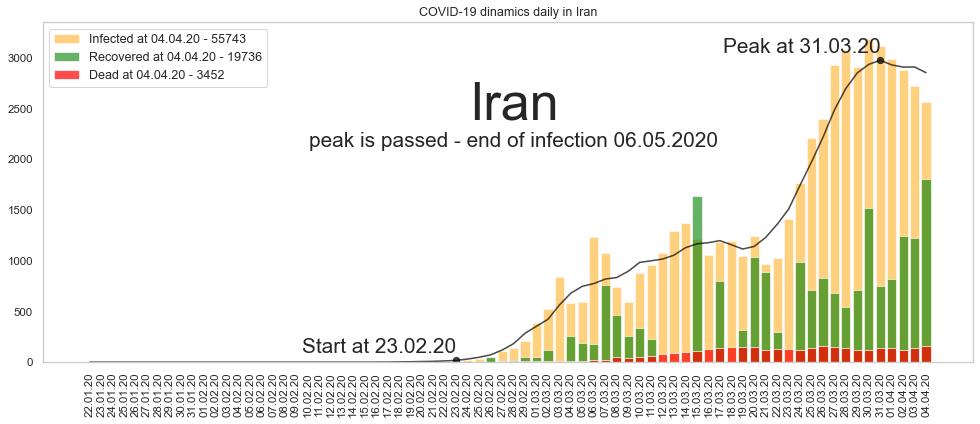
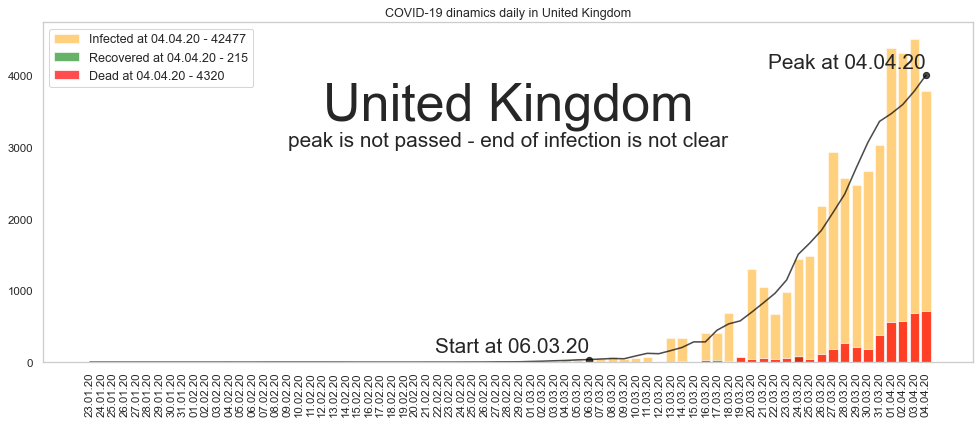

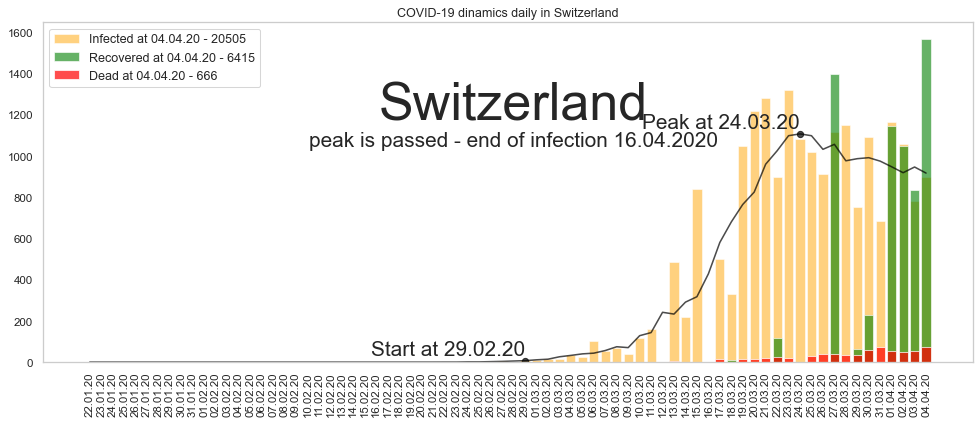
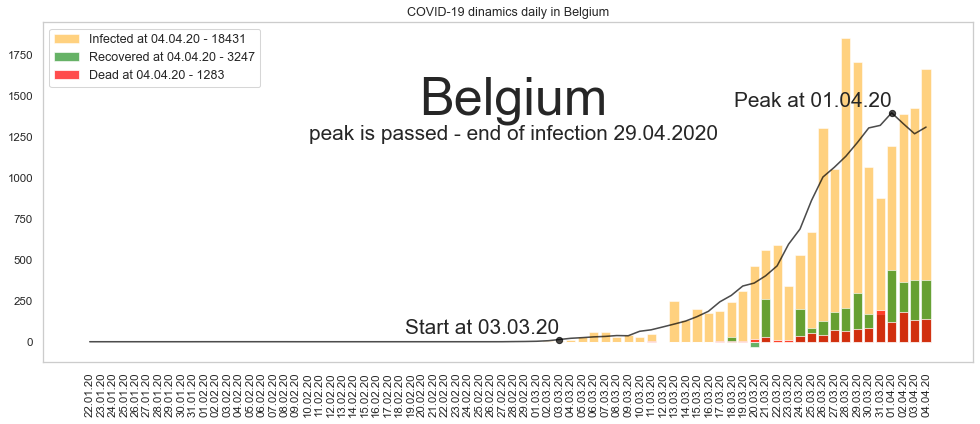
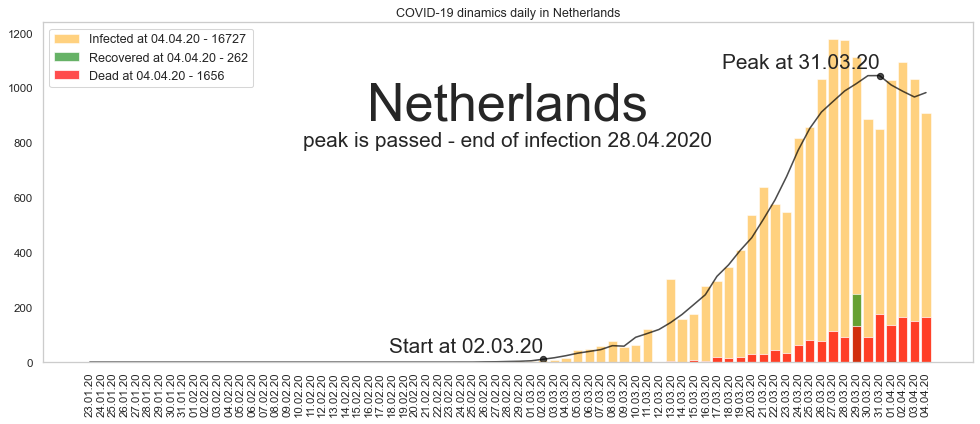
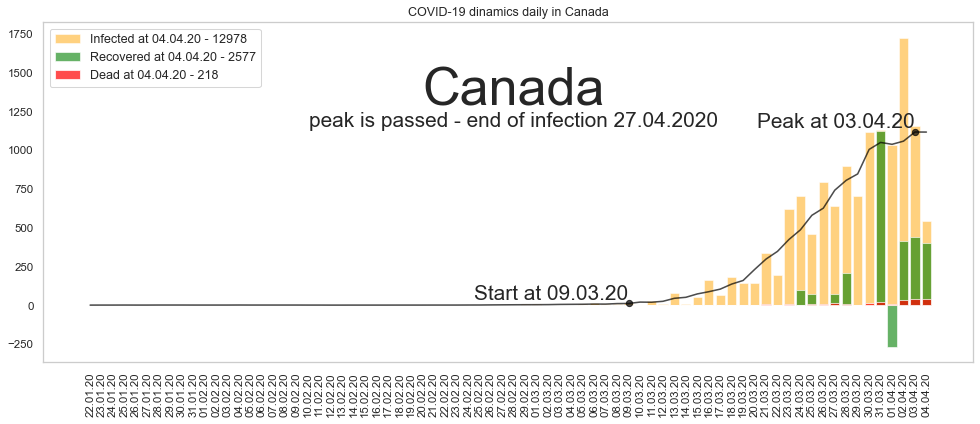
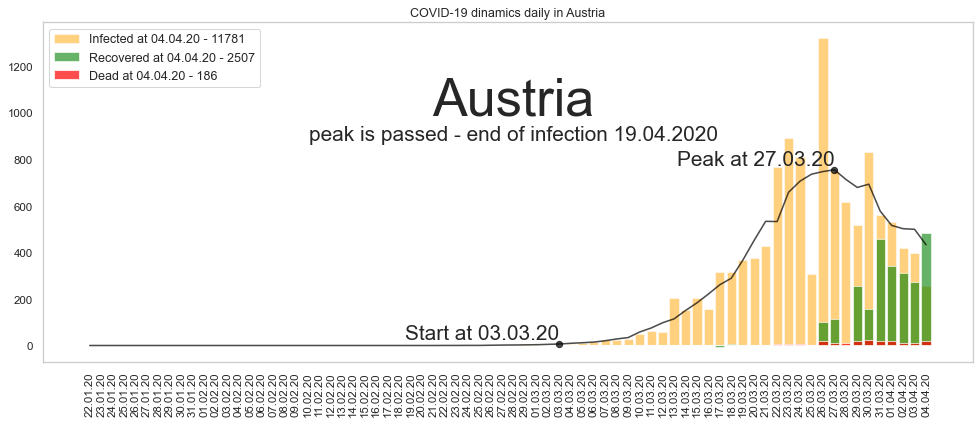

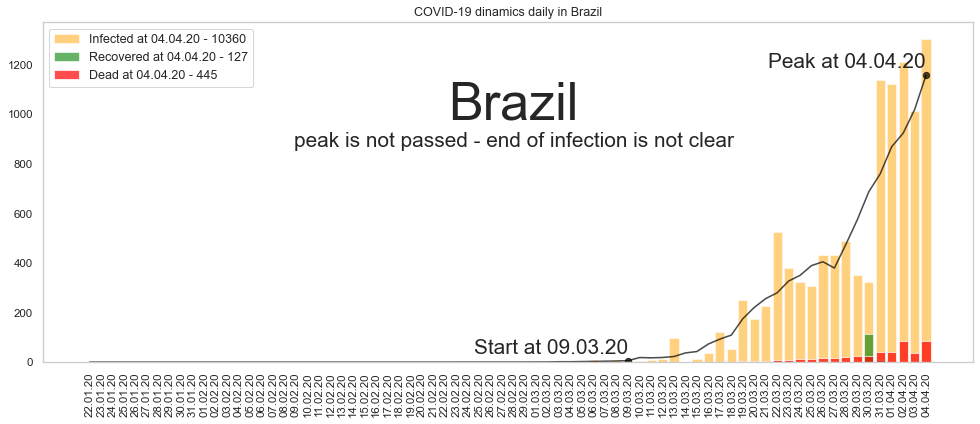
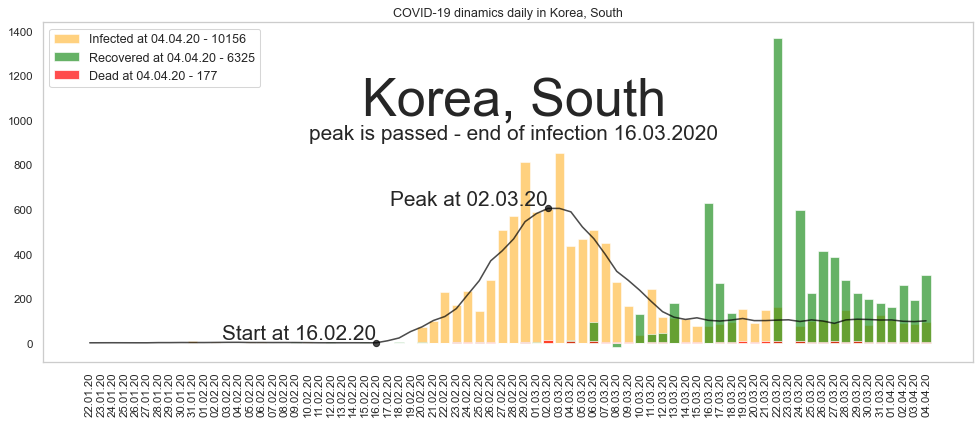
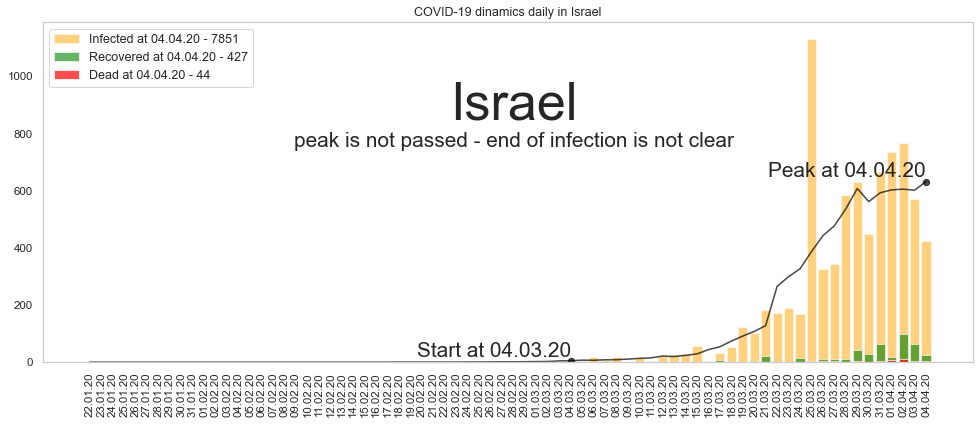
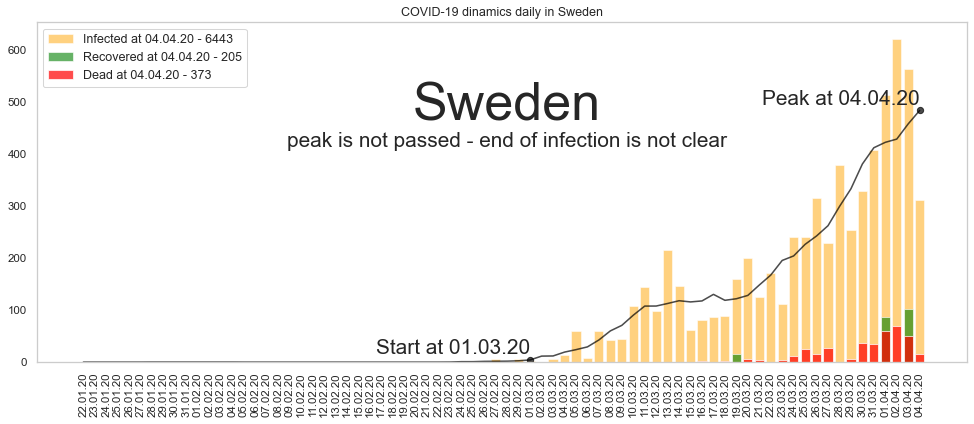
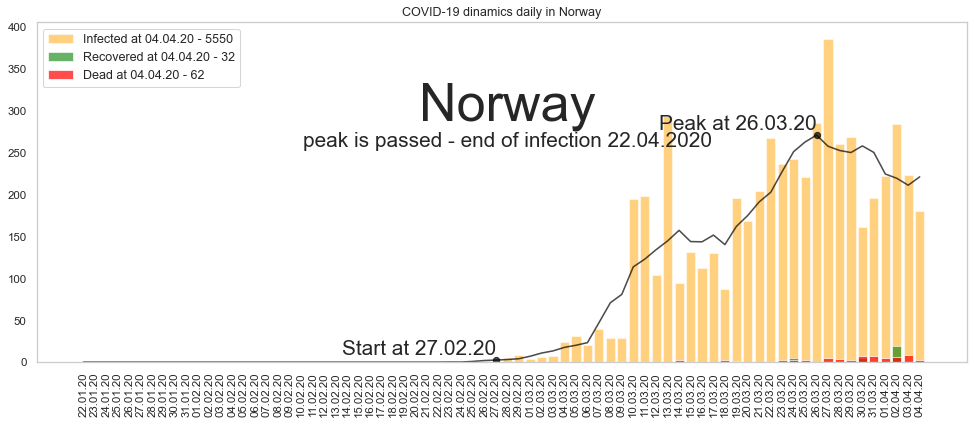
Автор: Валерий Курышев